 COVID.SI est un projet de science citoyenne pour lutter contre le SRAS-CoV-2 (virus de la COVID-19) par le calcul distribué.https://covid.si/en/
COVID.SI est un projet de science citoyenne pour lutter contre le SRAS-CoV-2 (virus de la COVID-19) par le calcul distribué.https://covid.si/en/
SiDock@home est une extension du projet COVID.SI basée sur BOINC pour engager la communauté du BOINC dans la recherche de nouveaux médicaments.
Informations sur le forum : https://forum.boinc-af.org/index.php/topic,8383.msg506899.html
À propos du projet
COVID.SI est un projet de science citoyenne pour lutter contre le SRAS-CoV-2 par le calcul distribué.
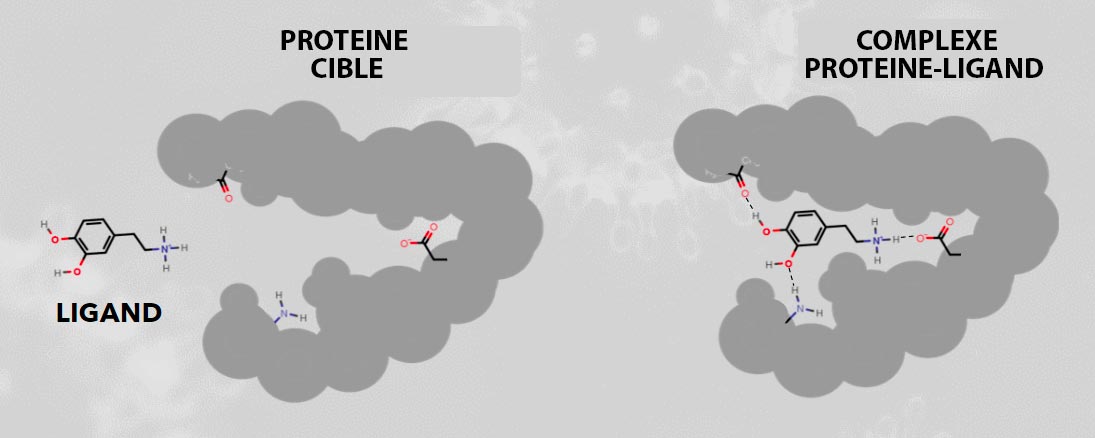
Dans notre projet, nous recherchons des ligands - de petites molécules capables de se lier avec succès à des cibles protéiques et de moduler un processus spécifique crucial pour la biochimie du virus. Ensemble, nous avons développé un logiciel qui peut être facilement installé sur votre ordinateur pour aider les participants à trouver le remède à l'ennemi invisible d'aujourd'hui. Sur la base de l'ancrage moléculaire, le ligand idéal doit être complémentaire en forme et en propriétés au site de liaison de la biomolécule cible. Cependant, la complémentarité des petites molécules n'est qu'une condition préalable à l'utilisation d'une molécule comme médicament.
La recherche de la bonne molécule est mieux illustrée en trouvant une petite aiguille dans une énorme botte de foin. C'est un problème beaucoup plus difficile en raison du nombre de structures moléculaires possibles. La quantité de toutes les structures moléculaires possibles est également appelée univers chimique - la taille de cet espace abstrait peut être estimée entre 10 et 80. Il existe de nombreuses galaxies dans cet univers chimique, et l'une des galaxies est une galaxie avec des molécules potentiellement pharmacologiquement actives. La méthode utilisée à cet effet est appelée dépistage virtuel. En principe, plus la partie de l'univers chimique que nous étudions est grande, plus la probabilité de trouver un remède potentiel au coronavirus est élevée.
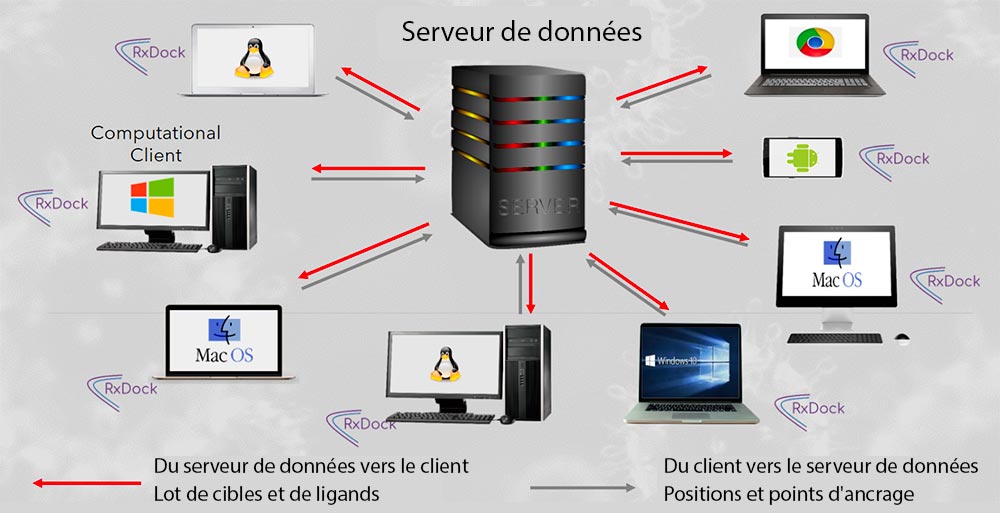
Vous pouvez aider avec votre ordinateur. Avec l'aide de BOINC, vous allez télécharger un sous-ensemble de composés sur votre ordinateur, examiner les composés dans le contexte de la cible étudiée et envoyer les résultats à un serveur où ils sont collectés pour une analyse ultérieure.
Présentation https://covid.si/p/1/en/
 COVID.SI
COVID.SI
projet de science citoyenne pour lutter contre le SRAS-CoV-2 par le calcul distribué
Dr. Črtomir Podlipnik, Dr. Marko Jukić et Sebastian Pleško
... et vous tous
 Si vous connaissez l'ennemi et que vous vous connaissez vous-même, vous ne devez pas craindre les résultats d'une centaine de batailles
Si vous connaissez l'ennemi et que vous vous connaissez vous-même, vous ne devez pas craindre les résultats d'une centaine de bataillesSun Tzu
Le SRAS-CoV-2 est un ennemi invisible que nous voulons connaître le mieux possible
Les coronavirus sont nos compagnons de tous les jours
- Les coronavirus sont nommés d'après les pointes de la couronne situées à leur surface
- Quatre grands sous-groupes de coronavirus : α, β, γ et δ (alpha, bête, gamma et delta)
- Les coronavirus humains ont été découverts au milieu des années 1960
- Les coronavirus humains communs de la sous-famille des Coronavirinae de la famille des Coronaviridae, qui sont souvent responsables des rhumes courants, sont : 229E (coronavirus alpha), NL63 (coronavirus alpha), OC43 (coronavirus bêta), HKU1 (coronavirus bêta)
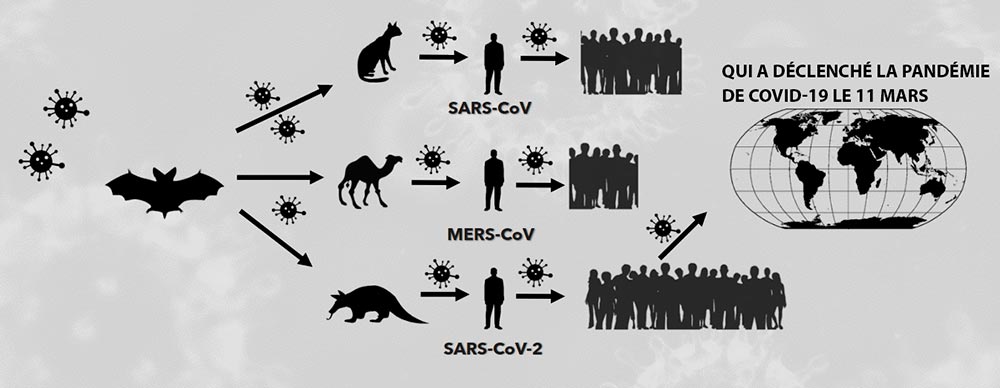
Parfois, les coronavirus, qui infectent généralement les animaux, peuvent être transmis à l'homme. Ces virus sont particulièrement dangereux car l'homme ne dispose pas encore des mécanismes de défense contre l'infection
Vidéo de l'organisation mondiale de la santé
Nouvelles récentes à propos de la pandémie de COVID-19
Le ZOO des protéines du SRAS-CoV-2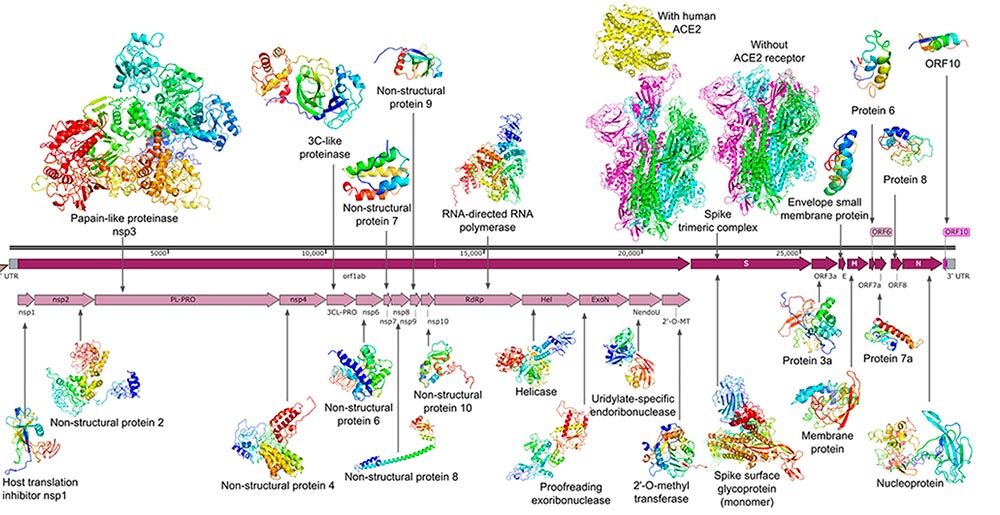
Modèles structurels 3D et annotation de fonction pour toutes les protéines codées par le génome du SARS-CoV-2. Les structures sont modélisées dans le laboratoire du professeur Zhang avec c-i-Tasser (https://zhanglab.ccmb.med.umich.edu/COVID-19/)
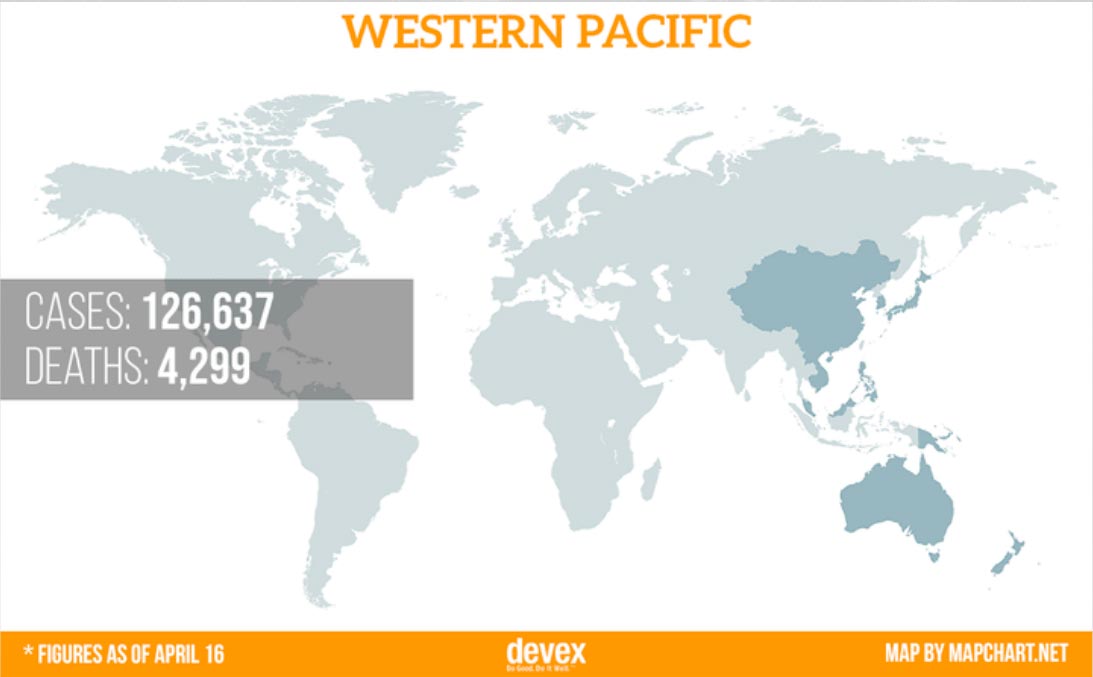
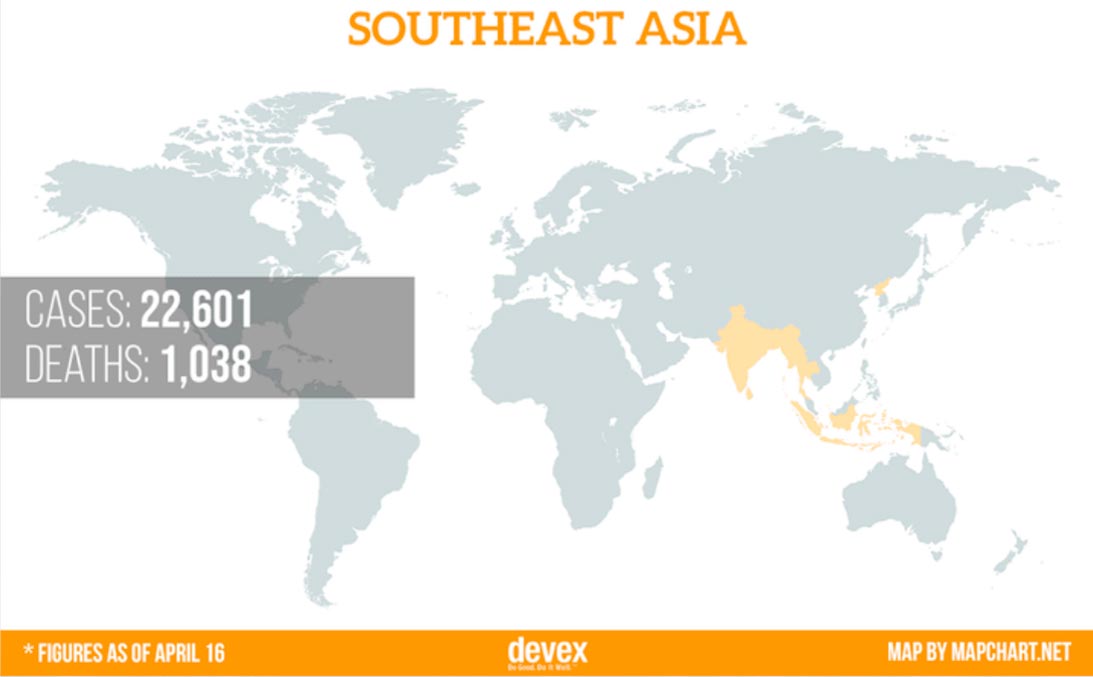
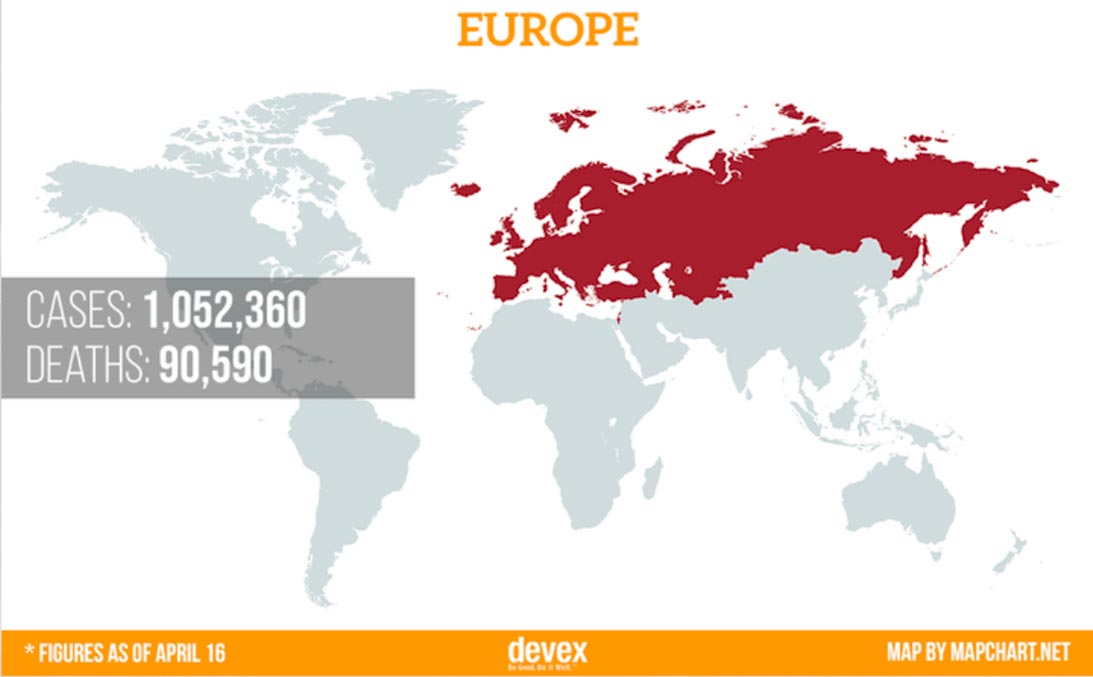
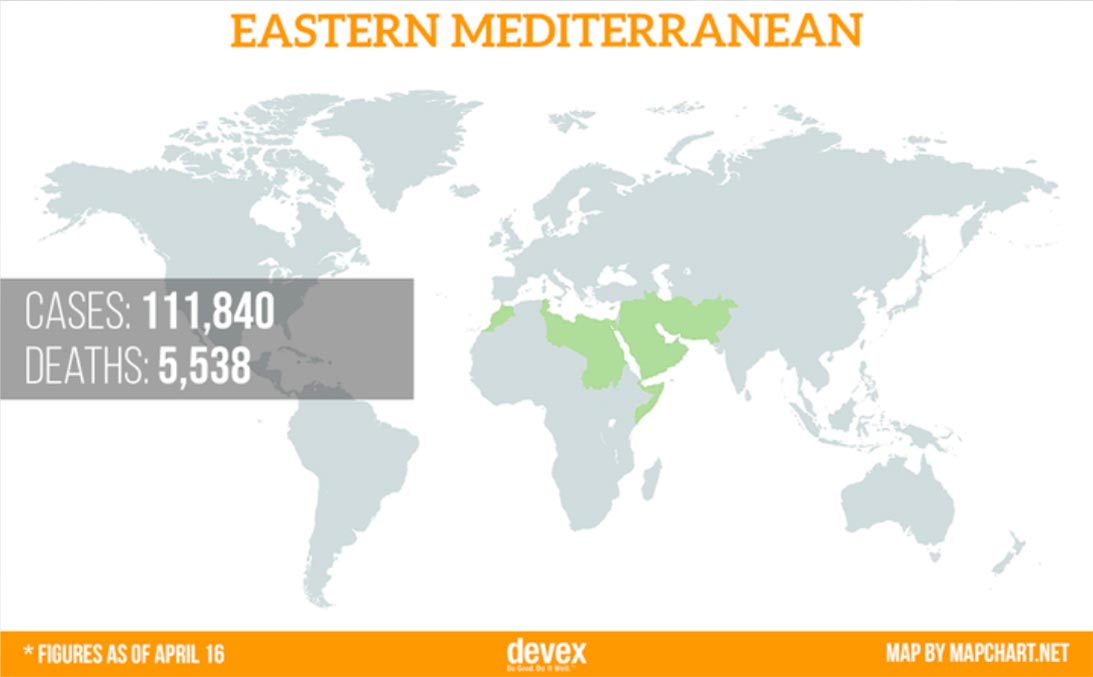
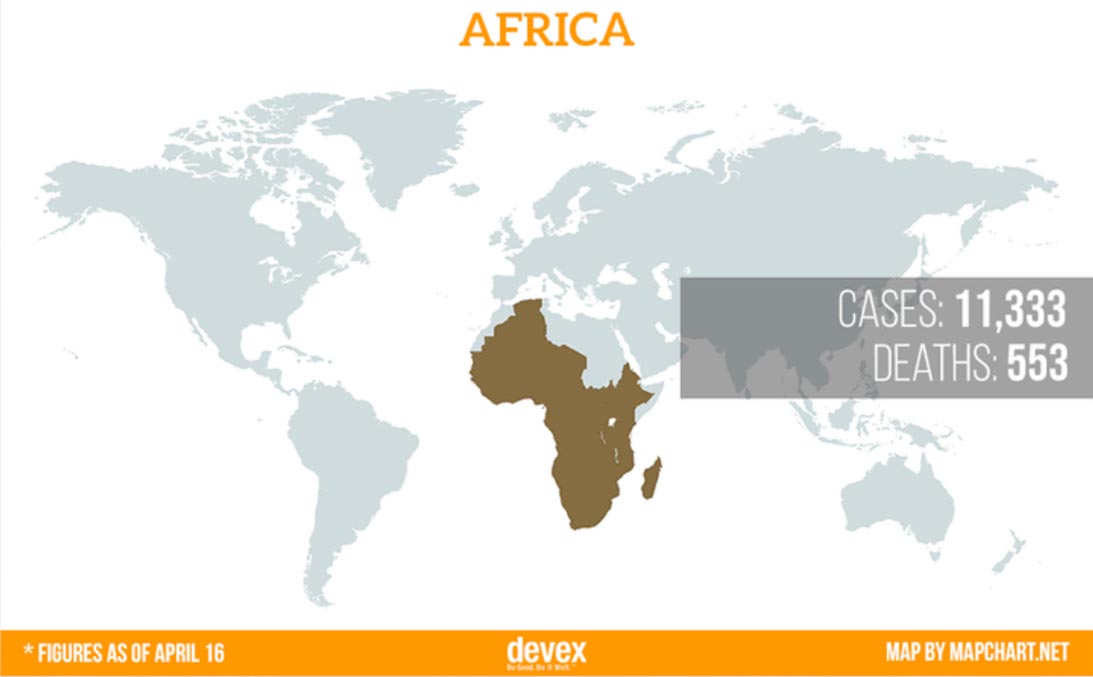
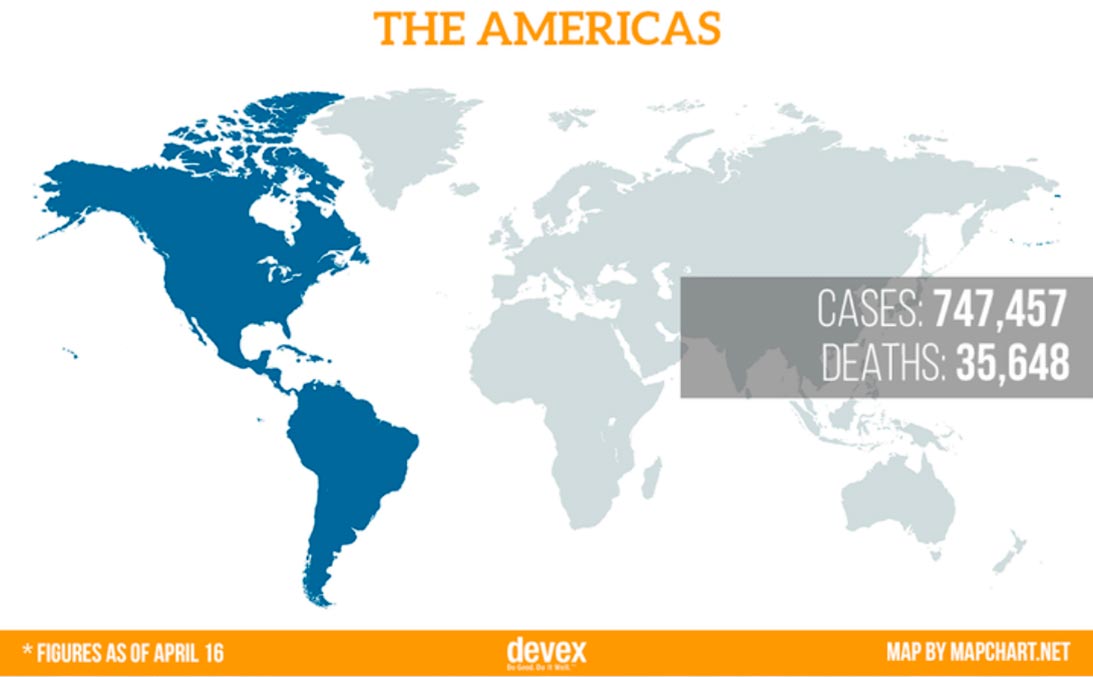
Le problème mondial de la santé
 |
 |
 |
||
 |
 |
 |
||
 |
||||
Un problème mondial nécessite une solution mondiale
Schéma de calcul distribué
Le système clé-serrure
L'action spécifique d'une enzyme sur un seul substrat peut être expliquée à l'aide d'une analogie avec une serrure et une clé, dont le premier postulat a été formulé en 1894 par Emil Fischer.
Dans cette analogie, la serrure est l'enzyme et la clé est le substrat. Seule la clé (substrat) de taille correcte s'insère dans le trou de serrure (site actif) de la serrure (enzyme).
http://chemistry.elmhurst.edu/vchembook/571lockkey.html
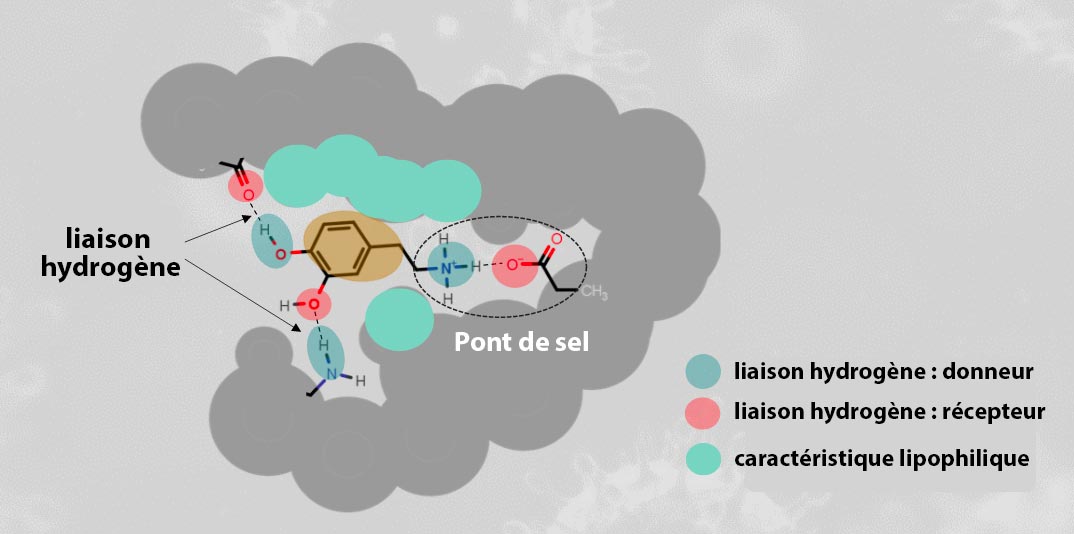
La connaissance de l'interaction entre un ligand et son hôte est cruciale pour comprendre la réponse biologique du lingand. La conception de médicaments implique la conception de molécules dont la forme et la charge sont complémentaires de la cible biomoléculaire avec laquelle elles interagissent et qui, par conséquent, se lient à elle.
Filtrage virtuel - Trouver la bonne clé pour la serrure

Le protocole de filtrage virtuel basé sur la structure, c'est comme chercher une clé qui déverrouille un cadenas dans une piscine olympique remplie de clés de toutes formes et de toutes tailles, sans garantie que la bonne clé se trouve dans la piscine.
L'espace chimique - source infinie de structures chimiques
 La stratégie sans tactique est le chemin le plus lent vers la victoire. La tactique sans stratégie est le bruit qui précède la défaite.
La stratégie sans tactique est le chemin le plus lent vers la victoire. La tactique sans stratégie est le bruit qui précède la défaite.Sun Tzu
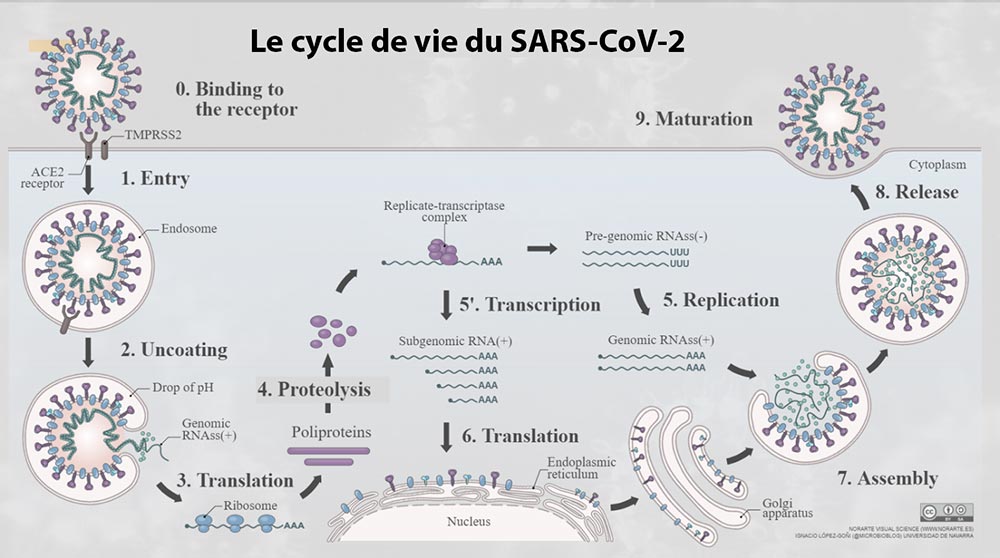
Stratégie 1 : Empêcher l'entrée du virus dans la cellule hôte
 Développement de molécules qui entravent les interactions entre la protéine à pointes du SRAS-CoV-2 et l'ACE2 ou d'autres récepteurs (en recherche active) :
Développement de molécules qui entravent les interactions entre la protéine à pointes du SRAS-CoV-2 et l'ACE2 ou d'autres récepteurs (en recherche active) :
- Conception d'anticorps monoclonaux humains neutralisants,
- Criblage d'une bibliothèque de petites molécules pour trouver des liants pour le domaine de liaison de reconnaissance des pointes,
- Criblage d'une bibliothèque de petites molécules pour trouver des inhibiteurs d'ACE2 (cible de l'hôte),
- Conception de petits peptides qui empêchent la liaison de la protéine de pointe au récepteur de l'hôte.
Autres récepteurs de l'hôte qui peuvent être impliqués dans l'entrée du virus : TMPRSS-2, GRP-78, CD-147
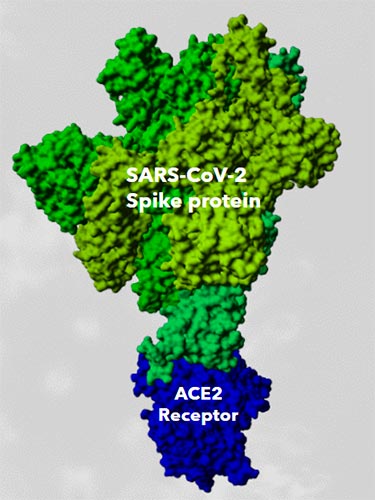
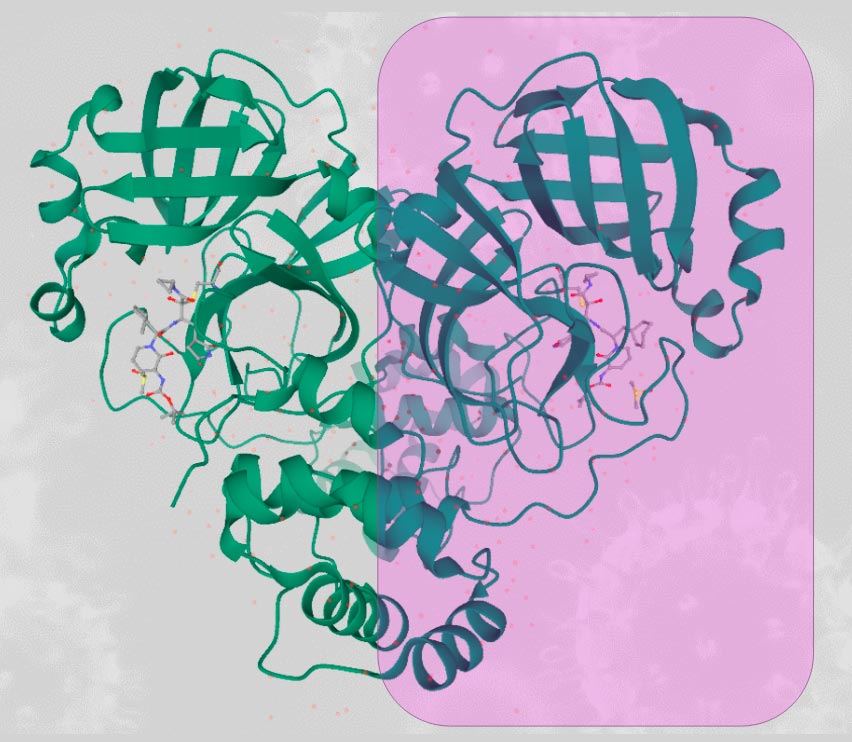
Le complexe entre la protéine S du SRAS-CoV-2 et le récepteur ACE2 de l'hôte, par le laboratoire Zhang, rendu avec YASARA.
Stratégie 2 : Empêcher le virus de s'échapper de l'endosome
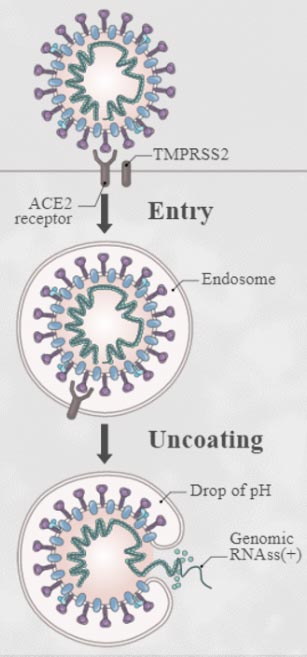
Agents lysosomotropes ciblant le pH endosomal/lysosomal
Thérapie avec la CQ, un médicament anti-paludisme bien connu, qui est un agent lysomotrope qui s'accumule dans les organites acides tels que les endosomes et les lysosomes et neutralise leur pH.
Inhibiteur de protéase endosomale-lysosomale
Inhibition des cathepsines, des protéases de cystéine endosomales et lysosomales qui jouent un rôle important dans la dégradation des protéines dans divers processus cellulaires, y compris la voie endocytaire et l'autophagie.
Inhibiteurs de l'endocytose à médiation par la clathrine
Recherche de nouveaux inhibiteurs de l'endocytose à médiation par la clathrine, l'un des principaux mécanismes d'entrée du virus dans les cellules hôtes.
Stratégie 3 : Désarmement de la protéase virale
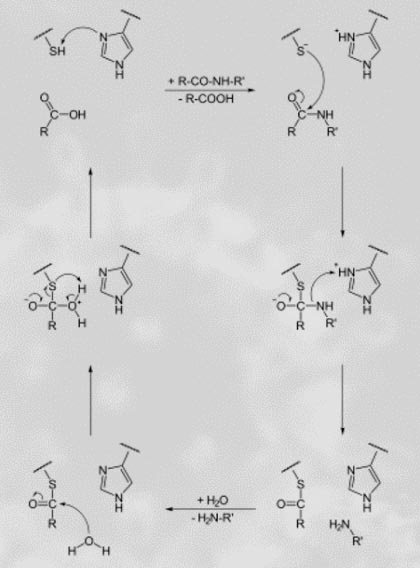
Le virus utilise ses protéases pour transformer les polyprotéines initiales en molécules protéiques fonctionnelles.
Actuellement, la protéase la plus étudiée est l'endopeptidase C30 ou protéinase de type 3C ( 3 CLpro). Il s'agit d'une cystéine protéase du clan PA, classification MEROPS C30.
Par conséquent, les voies d'inhibition de la protéase sont d'une importance capitale dans l'inhibition du cycle de vie du virus et les approches clés sont :
- Concevoir des petites molécules covalentes et
- Conception d'inhibiteurs non covalents à petites molécules
En parcourant l'univers des composés...
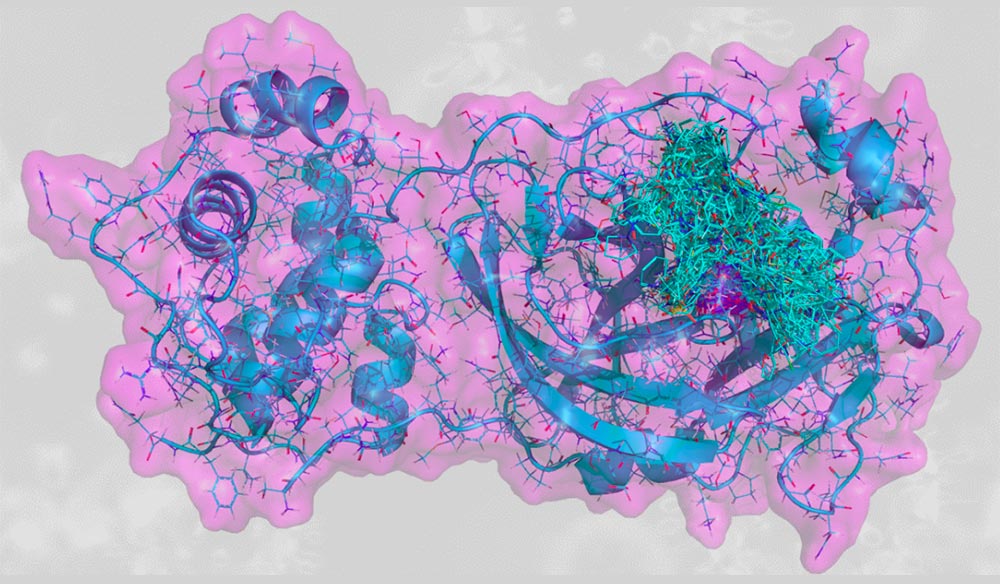
Les résultats des recherches en cours...
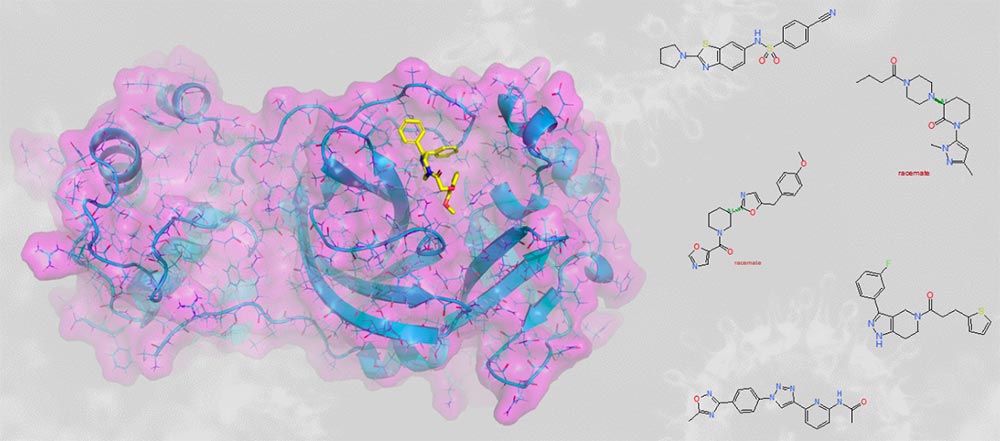
Des composés sont en cours d'évaluation biologique
Stratégie 4 : Inhibition d'autres cibles thérapeutiques potentielles telles que la RdRp, la protéine E et d'autres encore en recherche active
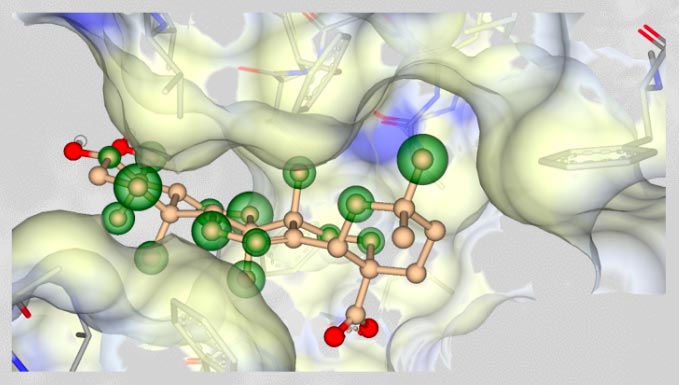 - La protéine virale est longue de 75 acides aminés et forme un pentamère, qui agit comme une protéine trans-enveloppe virale.
- La protéine virale est longue de 75 acides aminés et forme un pentamère, qui agit comme une protéine trans-enveloppe virale.
- Il fonctionne comme un canal ionique, très probablement pour le Na+, mais semble également être important pour la libération des virions des cellules.
- La suppression de la protéine virale E n'a pas tué le virus, mais a considérablement réduit sa croissance/diffusion/prolifération.
L'henderéganine liée au pentamère de la protéine E L'henderéganine semble être l'un des ligands les plus sûrs de notre étude. Elle est le principal constituant de l'extrait de feuille d'Hedera Helix. L'Hedera Helix est traditionnellement utilisé pour soulager les infections de type rhume/grippe.
Le projet COVID.SI remercie chaleureusement tous les participants pour leur soutien et leur aide !
Ensemble, nous sommes plus efficaces dans la lutte contre cette crise mondiale !

Merci de votre attention.
Equipe
- dr. Črtomir Podlipnik
- dr. Marko Jukić
- dr. Natalia Nikitina
- Sebastian Pleško
- Lorenzo Gilardoni
- Gašper Tomšič
- Žan Pevec
- Boštjan Laba
traduction d'après les sources : http://188.68.219.141/sidocktest/about.php - https://covid.si/p/1/en - https://covid.si/en/team/


