Je voudrais à nouveau réaffirmer mon entière gratitude à toutes les personnes qui ont contribué grâce à World Community Grid au projet "Help Defeat Cancer" (HDC). Comme dans la plupart des recherches scientifiques, notre équipe a commencé le projet par une idée innovante qui a ensuite été renforcé par des fondements scientifiques, mais celà requiert une recherche systématique pour démontrer sa validité. Dans notre cas, nous avons opté pour une approche analytique à grande échelle pour caractériser les shémas d'expression des protéines au travers des spécimens de tissu qui pourraient permettre de classifier fidèlement les sous-catégories et les étapes de la progression du cancer du sein, du côlon, de la tête et du cou.
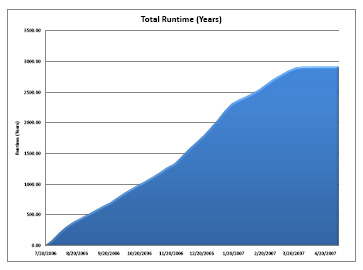
World Community Grid a alloué plus de 2 900 années de calcul au projet HDC, ce qui nous a permis de démontrer la faisabilité de l'utilisation des signatures spectrales et spatiales pour caractériser les shémas d'expression des différents spécimens de cancer. En outre, nous avons été en mesure de démontrer que la bibliothèque des biomarqueurs ne souffre d'aucune contrainte spatiale et peut donc être utilisée pour analyser les structures multi-niveaux, c'est à dire au niveau du disque, du tissu, et de la tumeur. Pendant le même temps nous avons développé un nouveau descripteur de régions covariantes présenté comme ayant de meilleures performances dans la classification des tumeurs ainsi qu'un cadriciel de segmentation qui pourra sûrement représenter les différentes catégories de cellules et de tissus même lorsqu'elles se présentent sous un angle, un éclairage, et une échelle différente.
Suite à l'amélioration des résultats expérimentaux obtenus durant le projet "Help Defeat Cancer", nous avons addressé une nouvelle demande de subvention à l'institut national de la santé (NIH) pour l'attribution d'une aide financière intitulée, « recherche, filtrage et catégorisation d'images pour l'analyse comparative des schémas d'expression des matrices tissulaires ». L'objectif central de cette demande est de mettre en place des systèmes interactifs d'aide à la décision clinique pour permettre aux chercheurs et aux médecins d'analyser et de classifier automatiquement les spécimens de cancer afin d'améliorer l'exactitude du diagnostic et du pronostic médical. La preuve du concept du système proposé a été ammené par l'utilisation de la bibliothèque de référence des shémas d'expression des biomarqueurs qui a été produite dans le cadre du projet HDC.
Nous avons récemment été informé que la proposition de recherche que nous avons soumis au NIH a reçu un score de 3,9 percentiles (les percentiles indiquent un classement relatif entre les projets candidats). Pour examiner la performance des nouvelles technologies et des outils informatiques développés au cours de ces 4 années de fonctionnement du projet, un réseau virtuel sera mis en place dans différents emplacements stratégiques situés à l'institut du Cancer du New Jersey (CINJ), à l'Université Columbia (CU), à l'Université de l'État de l'Ohio (OSU), l'Université Rutgers (RU), et l'Ecole médecine de Pennsylvanie (UPenn). Le logiciel et les technologies sous-jacentes développés à partir de ce projet seront rendus disponibles aux chercheurs par le biais des scientifiques et de la communauté des cancérologues pour une utilisation qui viendrait en appui aux décisions cliniques, à la recherche fondamentale et aux découvertes.
Bien que cette phase du projet "Help Defeat Cancer" touche à sa fin, je reste convaincu que ce n'est que le début d'un nouveau commencement. Ceci vient d'être confirmé alors que nous nous approchons du début du nouveau projet financé par le NIH et que nous commençons une nouvelle collaboration avec les scientifiques du centre de recherche T.J. Watson d'IBM. L'objectif central du nouveau projet avec le soutien d'IBM est de progresser dans nos travaux préliminaires afin de développer un ensemble de méta-classificateurs multi-modaux capablent d'évaluer simultanément le profil clinique à partir des images des matrices tissulaires, du génome, et du protéome des patients afin d'augmenter l'exactitude dans la planification de la détection, du traitement et de la thérapie.
12 Juin 2007 - Dernières nouvelles en provenance de l'Institut du Cancer du New Jersey
Le laboratoire d'imagerie et d'informatique biomédicale (CBII) va soutenir un certain nombre de nouvelles publications :
-
Lin Yang, Peter Meer, David J. Foran. Segmentation pixel par pixel de catégories multiples par l'utilisation d'un histogramme d'estimation non paramétrique par noyau (Mean Shift). Conférence de l'IEEE sur la vision cognitive et les modèles de reconnaissance informatique. Publication retenue pour être présentée lors de cette conférence internationale qui se tiendra à Minneapolis dans le Minnesota en Juin 2007.
-
Lin Yang et David J. Foran. Approche variationnelle pour l'oclusion partielle d'une segmentation d'image par l'utilisation d'un trait épais pour un bon équilibrage et d'une estimation semi-paramétrique de la densité. Conférence de l'IEEE sur le traitement de l'image. Publication retenue pour être présentée lors de cette conférence internationale qui se tiendra à San Antonio au Texas en Septembre 2007.
-
Lin Yang, Peter Meer, Lauri A. Goodell, Michael D. Feldman, et David J. Foran. Analyse du cancer du sein à grande échelle sur la grille. Conférence sur l'imagerie médicale et l'intervention assistée par informatique. Publication retenue pour être présentée lors de cette conférence internationale qui se tiendra à Brisbane en Australie au mois d'Octobre 2007.
-
Bonnie H. Hall, Wenjin Chen, et David J. Foran. Cadriciel à utilisation clinique pour quantifier et classifier des spécimens colorés par immunohistochimie. Conférence sur l'imagerie médicale et l'intervention assistée par informatique. Publication retenue pour être présentée lors de cette conférence internationale qui se tiendra à Brisbane en Australie au mois d'Octobre 2007.
-
Gabriela Niculescu, John L. Nosher, David J. Foran. Observation d'un foie suite à des études comparatives sur la réponse de la tumeur à une ablation par radio fréquence. Conférence IEEE de l'association d'ingénierie médicale et biologique. Publication retenue pour être présentée lors de cette conférence internationale qui se tiendra à Lyon, en Août 2007.
Quelques vidéos
- Guide imagé et système de soutien à la décision (.rm, 9,53 Mo, 2min56)
- Télémicroscopie partagée (.rm, 5,3 Mo, 2min30)
- Analyse de cellules autonomes (.rm, 3,3 Mo, 1min25)