Avec la récente épidémie de COVID-19, Rosetta@home a été utilisé pour prédire la structure des protéines importantes pour la maladie ainsi que pour produire de nouvelles mini-protéines stables à utiliser comme thérapies et diagnostics potentiels, comme celui affiché ci-dessous qui est lié à une partie de la protéine de pointe SARS-CoV-2.
Pour aider nos recherches, nous sommes heureux d'annoncer une nouvelle mise à jour de l'application, et grâce à l'aide de la communauté de développement Arm, y compris Rex St. John, Dmitry Moskalchuk, David Tischler, Lloyd Watts et Sahaj Sarup, nous sommes ravis de inclure la plate-forme Linux-ARM. Avec cette mise à jour, nous continuerons à fabriquer des liants protéiques pour le SRAS-CoV-2 et les cibles connexes en utilisant la dernière source Rosetta.
Merci aux bénévoles de R @ h pour votre soutien continu à ce projet. Vos heures CPU sont utilisées non seulement pour modéliser avec précision les structures de protéines importantes, mais également pour en concevoir de nouvelles. Unissons-nous et combattons COVID-19!
Plus de détails seront disponibles dans la discussion de cet article.
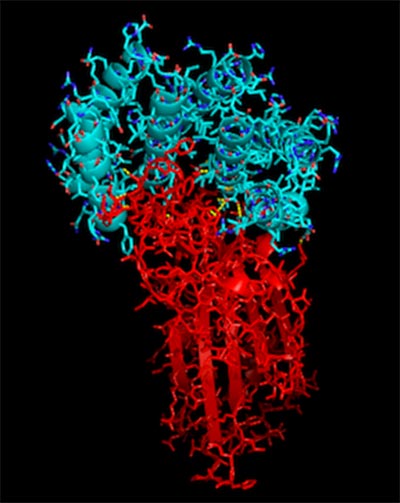
La principale façon dont les protéines interagissent les unes avec les autres est de se coller les unes aux autres. Comme vous l'avez peut-être vu dans l'application graphique Rosetta@home, les protéines se présentent sous toutes les formes et tailles. Pour cette raison, la plupart des protéines ne collent pas au hasard les unes aux autres, mais collent plutôt très spécifiquement à une poignée d'autres protéines. Par exemple, la protéine de pointe virale du SRAS-CoV-2 colle à la protéine ACE2 humaine, ce qui permet au virus de pénétrer dans la cellule.
L'IPD a travaillé dur pour améliorer la capacité de concevoir de telles interactions de liaison. Ce processus commence par la création d'un ensemble de protéines d'échafaudage qui n'ont d'autre but que de se replier précisément en une structure atomique. Ces échafaudages sont ensuite ancrés sur une protéine cible d'intérêt et leurs surfaces conçues pour compléter parfaitement la cible. Enfin, les conceptions sont notées, filtrées et testées pour la liaison en laboratoire.
Nous allons maintenant utiliser Rosetta@home pour effectuer l'étape de conception de la surface. L'amarrage et le filtrage sont rapides, mais en réalité, la conception de protéines est lente. Nous utiliserons les énormes quantités de puissance de calcul disponibles sur Rosetta@home pour échantillonner chaque acide aminé à chaque position à l'interface. Nous choisirons ensuite les meilleures combinaisons d'acides aminés en utilisant le recuit simulé et Monte-Carlo. L'échantillonnage est la clé de ce processus et c'est pourquoi nous nous tournons vers Rosetta@home.
Alors, rejoignez-nous dans les semaines à venir alors que nous fabriquons des liants pour le SRAS-CoV-2 et les protéines associées. Nous continuerons à faire de la prédiction de structure et de la conception d'échafaudage, car ils sont absolument essentiels à la science des protéines. Mais restez attentif aux cas de conception d'interface, car quelqu'un pourrait concevoir le prochain remède COVID-19.
Et j'espère que vous resterez une fois la pandémie terminée. Nous ne pouvons concevoir des liants comme celui-ci que parce que nous travaillons dur sur le problème depuis des années. Il y a cependant encore un long chemin à parcourir. L'amélioration de la science prend du temps et de la puissance informatique, nous espérons donc que vous vous joindrez à nous pour cette aventure passionnante.
Brian Coventry
03/04/2020
https://boinc.bakerlab.org/rosetta/forum_thread.php?id=13702


