Récapitulatif
L'équipe Mapping Cancer Markers discute du passé (cancer du poumon), du présent (cancer de l'ovaire) et du futur (sarcome) du projet dans cette mise à jour complète.
Contexte
Le projet Mapping Cancer Markers (MCM) a été conçu pour identifier les marqueurs associés à divers types de cancer et, en affinant le processus d'identification de ces marqueurs, identifier plus efficacement ces biomarqueurs pour d'autres maladies. Notre objectif était d'analyser plusieurs ensembles de données sur le cancer afin d'identifier des biomarqueurs potentiels pour ces cancers, ce qui pourrait éventuellement aider les scientifiques et les médecins à détecter les cancers plus tôt et à créer des traitements personnalisés. Les trois premiers ensembles de données du plan MCM sont le poumon, l'ovaire et le sarcome, représentant le passé, le présent et l'avenir du MCM. Le traitement pulmonaire est terminé. Le traitement des marqueurs ovariens est en cours, mais presque terminé. Nous nous préparons maintenant à passer au sarcome.
Le traitement d'un ensemble de données sur World Community Grid au cours des mois et des années produit une énorme quantité de données, et ces données ne sont pas directement utilisables, mais doivent ensuite être rassemblées, filtrées et analysées de différentes manières. Nous nous sommes concentrés sur cette étape de post-traitement dans notre laboratoire.
Dans cette mise à jour, nous discuterons principalement de certains des travaux effectués avec l'ensemble de données pulmonaires traitées, mais d'abord, nous jetterons un rapide coup d'œil sur l'avenir.
Derniers préparatifs pour l'ensemble de données sur le sarcome
Le prochain jeu de données sur le sarcome sera le jeu de données le plus complexe de MCM à ce jour. Il contient des biomarqueurs potentiels tirés de sources multiples: mesures de l'ARN, de l'ADN et de l'activité protéique, mutations et autres modalités biologiques.
Avec ces informations détaillées sur chaque échantillon de l'ensemble de données, il a fallu un certain effort pour réduire l'ensemble de données et la taille des résultats à des niveaux pratiques. Nous testons actuellement les unités de travail de notre projet de jeu de données et planifions les travaux.
Une future mise à jour annoncera le lancement d'une nouvelle phase du projet MCM, axée sur le sarcome et fournira plus de détails.
Résultats de l'ensemble de données pulmonaires
Les biomarqueurs de l'ensemble de données pulmonaires MCM mesurent l'activité de milliers de gènes. Collectivement, ces biomarqueurs couvrent la majeure partie du génome humain. La majorité du travail pulmonaire MCM traité sur les signatures sondées World Community Grid tirées au hasard de l'ensemble des biomarqueurs. Une deuxième phase plus courte du poumon MCM a attiré des signatures à partir de sous-ensembles optimisés de ces biomarqueurs.
La contribution des cycles de calcul au projet a été extraordinaire. Les membres du World Community Grid ont traité 4,5 billions de signatures de cancer du poumon candidat dans la phase principale du poumon MCM, 220 milliards dans une phase expérimentale initiale et 1,6 billion de signatures dans la phase d'optimisation.
Nous discuterons de certaines découvertes de la phase principale du poumon MCM dans cette mise à jour.
La question de la taille de la signature
Poumon MCM ont étudié des signatures de plusieurs tailles. Les tailles variaient de 5 biomarqueurs à 100, avec le plus grand accent sur les signatures dans la gamme de 10-20 biomarqueurs. Pour qu'une signature cancéreuse réussisse en utilisation clinique, la taille de la signature est un compromis entre la puissance diagnostique, la complexité et le coût. Chaque biomarqueur peut potentiellement ajouter des informations de diagnostic à une signature, ce qui augmente la précision, mais trop de biomarqueurs peuvent également ajouter du bruit et augmenter inutilement les coûts et la complexité pour une utilisation pratique en clinique.
La figure ci-dessous montre l'effet de la taille de la signature sur la précision maximale. Pour presque toutes les tailles, une signature construite à partir de biomarqueurs choisis au hasard aura une faible précision, mais en testant suffisamment de telles signatures, puis en examinant la précision de la fraction supérieure (disons, le 0,01% le plus élevé), nous voyons l'effet produit par taille de la signature. Des signatures soigneusement conçues devraient atteindre la même précision en utilisant moins de biomarqueurs.
Figure 1A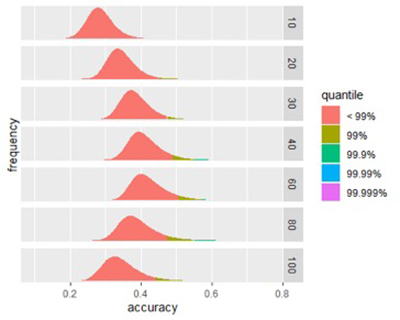
Figure 1B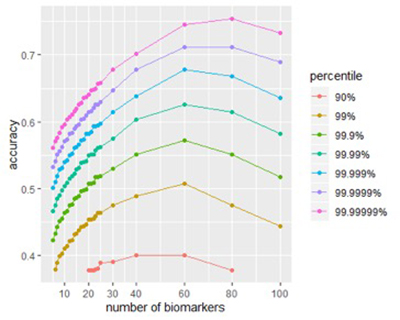
Figures 1A et 1B: La taille affecte la précision potentielle d'une signature. (A) La distribution des scores des signatures réussies de différentes tailles. (B) Un examen plus approfondi de l'effet de la taille de la précision du score. La précision maximale de se trouve dans les signatures entre 40 et 80 biomarqueurs.
Quels biomarqueurs ont le plus de succès ?
Dans la phase principale du poumon MCM, les signatures ont été construites à partir de biomarqueurs choisis au hasard dans l'ensemble de données. En tant que tel, chaque biomarqueur avait une chance égale d'apparaître dans chaque nouvelle signature. Cela ne signifie pas, cependant, que tous les biomarqueurs sont également utiles - comme nous l'avons dit ci-dessus, une signature aléatoire aura très probablement une faible précision. Si, cependant, nous prenons seulement la fraction la plus précise des signatures et voyons quels biomarqueurs elles contiennent, nous constatons que quelques biomarqueurs apparaissent fréquemment et que les autres sont relativement rares. (Nous pouvons même remarquer des modèles de manière à ce que certains groupes de biomarqueurs apparaissent ensemble, comme nous l'avons expliqué dans une mise à jour précédente.) Nous pouvons alors déterminer l'efficacité ou l'utilité de chaque biomarqueur en fonction de la fréquence à laquelle il apparaît dans ces signatures supérieures.
Figure 2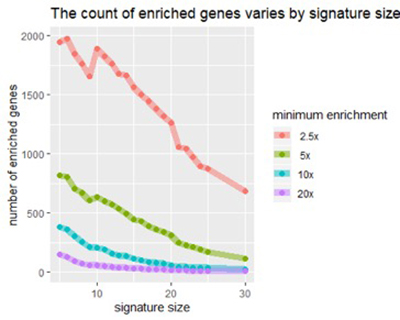
Figure 2: À mesure que la taille de la signature augmente, nous constatons une diminution du nombre de gènes enrichis par n'importe quel facteur (par exemple, 5 fois au-dessus de la normale).
Après avoir analysé l'ensemble complet des résultats pulmonaires MCM, nous pouvons confirmer un effet que nous avions remarqué dans des études préliminaires antérieures: l'efficacité de chaque biomarqueur dépend de la taille de la signature, affectant chaque biomarqueur différemment. La figure ci-dessous illustre l'effet de certains des biomarqueurs les mieux classés.
Figure 3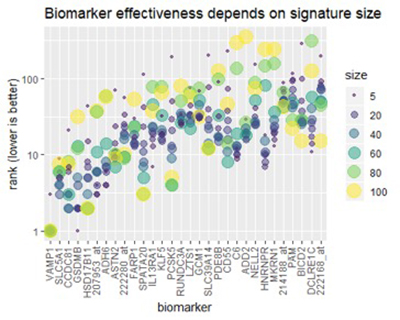
Figure 3: La taille de la signature du cancer du poumon détermine l'utilité d'un biomarqueur.
À mesure que la taille de la signature augmente, les biomarqueurs individuels peuvent devenir plus ou moins efficaces.
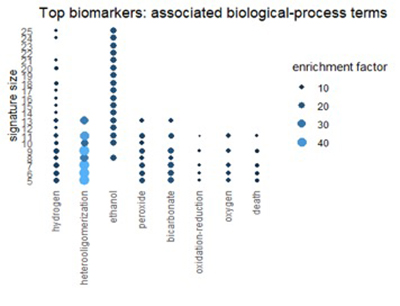
Enrichissement des voies parmi les meilleurs biomarqueurs
Pour obtenir une vue de plus haut niveau des biomarqueurs découverts dans l'ensemble de données pulmonaires, nous les avons examinés du point de vue de la voie. Une voie est un groupe de gènes qui coopèrent pour remplir la même fonction biologique. Nous avons introduit des listes de biomarqueurs parmi les 1% les plus performants dans la base de données pathDIP de notre laboratoire [1], [2]. pathDIP est une base de données complète et intégrée des voies connues (cascades de signalisation), et étant donné une liste de gènes, il trouvera toutes les voies associées à n'importe quel gène dans la liste. Plus utilement, il mesurera l' enrichissement de chaque voie dans votre liste de gènes - le degré auquel la voie a une connexion supérieure à la moyenne à votre liste. En utilisant une telle analyse, nous visons à trouver une interprétation biologiquement significative de nos biomarqueurs identifiés.
La figure ci-dessous montre les résultats de pathDIP.
Figure 4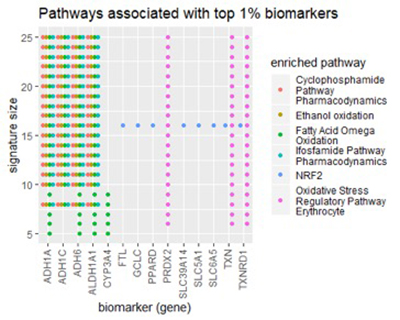
Sur un grand nombre de tailles de signature, pathDIP a toujours trouvé cinq voies enrichies dans nos listes de gènes:
- Voie cyclophosphamide, pharmacodynamique
- Voie de l'ifosfamide, pharmacodynamique
- Oxydation à l'éthanol
- Oxydation des acides gras oméga
- Voie de régulation du stress oxydatif (érythrocyte)
Les cinq voies enrichies sont liées au métabolisme, ce qui signifie la dégradation des produits chimiques dans le corps. Curieusement, les deux premières voies concernent spécifiquement le métabolisme des médicaments de chimiothérapie, le cyclophosphamide et l'ifosfamide. Les trois derniers concernent soit l'oxydation, soit la prévention du stress oxydatif (radicaux libres) dans les globules rouges.
Utilisation de Gene Ontology Resource pour décrire les meilleurs biomarqueurs
Nous pouvons obtenir une vue connexe à partir de la ressource d'ontologie génique (GO). GO classe chaque gène selon trois perspectives différentes: processus biologique, fonction moléculaire et composante cellulaire. Les chiffres ci-dessous montrent les termes des catégories GO qui apparaissent fréquemment dans les 1% des biomarqueurs les plus importants.
Figure 6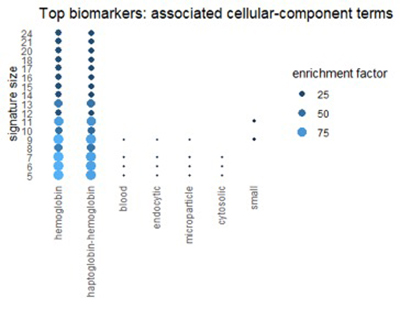
Figure 7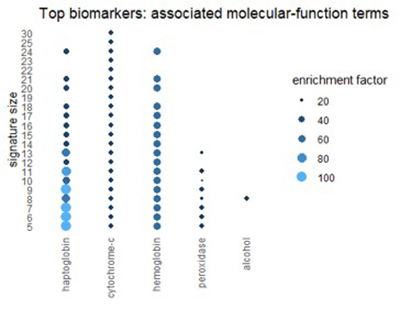
Beaucoup de termes reflètent les thèmes trouvés dans les voies: l'oxydation, l'alcool et la chimie des globules rouges.
Perspectives d'avenir
Nous sommes en train d'étendre et de combiner plusieurs analyses supplémentaires des données pulmonaires de la phase principale et des analyses substantielles des résultats pulmonaires de la deuxième phase. Après cela, les données ovariennes attendent. Pour l'ovaire, certaines des mêmes techniques s'appliqueront, mais certaines devront être adaptées et d'autres devront être développées.
Bref, le projet MCM nous occupera longtemps. En attendant, nous tenons à vous remercier pour votre intérêt et pour votre généreux don de puissance de calcul à ce projet et à d'autres projets du World Community Grid. Nous fournirons des mises à jour plus fréquemment maintenant.
Résultats supplémentaires
Au cours des deux dernières années, nous avons publié plusieurs manuscrits originaux et plusieurs applications à l'aide de nos outils et programmes, dont beaucoup nous avons utilisé pour augmenter la valeur des analyses MCM:
- PMID: 31583635 Kennedy, S., Jarboui, MA, Srihari, S, Raso, C, Bryan, K, Dernayka, L, Charitou. T, Bernal-Llinares, M, Herrera-Montavez, C, Krstic, A, Matallanas, D, Kotlyar, M , Jurisica, I , Curak, J, Wong, V, Stagljar, I, LeBihan, T, Imrie, L, Pillai, P, Lynn, M, Fasterius, E, Szigyarto, C. AK, Breen, J, Kiel, C, Serrano, L, Rauch, N, Rukhlenko, O, Kholodenko, B, Iglesias-Martinez, L, Ryan, C, Pilkington, R, Cammareri, P, Sansom, O, Shave, S, Auer, M, Horn, N, Klose, F, Ueffing, M, Boldt, K, Lynn, D, Kolch, W, Rewiring extensif du EGFR Network in Colorectal Cancer Cells Expressing Transforming Levels of KRASG13D, Nat Commun , 2019. Sous presse.
- Enfield, KSS, Marshall, EA, Anderson, C., Ng, KW, Rahmati, S , Xu, Z. Fuller, M., Milne, K., Lu, D., Shi, R., Rowbotham, DA, Becker -Santos, DD, Johnson, FD, anglais, JC, MacAulay, CE, Lam, S., Lockwood, WW, Chari, R., Karsan, A., Jurisica, I., Lam, WL, Suppresseur de tumeur épithélial ELF3 est un oncogène amplifié spécifique à la lignée dans l'adénocarcinome pulmonaire, Nat Commun , 10 (1): 5438, 2019. doi: 10.1038 / s41467-019-13295-y
- Rahmati, S., Abovsky, M., Pastrello, C., Kotlyar, M., Lu, R., Cumbaa, CA , Rahman, P., Chandran, V. et Jurisica, I. pathDIP 4: An Extended Pathway Annotations et ressource d'analyse d'enrichissement pour les humains, les organismes modèles et les espèces domestiquées, Nucl Acids Res , sous presse. 2019. https://doi.org/10.1093/nar/gkz989
- Holzinger A, Haibe-Kains B, Jurisica I. Pourquoi l'imagerie seule ne suffit pas: intégration basée sur l'IA d'imagerie, d'omique et de données cliniques, Eur J Nucl Med Mol Imaging . 15 juin 2019 doi: 10.1007 / s00259-019-04382-9.
- Monette A, Bergeron D, Ben Amor A, Meunier L, Caron C, Mes-Masson AM, Kchir N, Hamzaoui K, Jurisica I , Lapointe R. L'enrichissement immunitaire des biopsies de base du cancer du poumon non à petites cellules pour le profilage multiplexe définit le pronostic combinaisons de points de contrôle immunitaire pour la stratification des patients, J Immunother Cancer, 7 (1): 86, 2019. doi: 10.1186 / s40425-019-0544-x
- Monette A, Morou A, Al-Banna NA, Rousseau L, Lattouf JB, Rahmati S, Tokar T , Routy JP, Cailhier JF, Kaufmann DE, Jurisica I , Lapointe R. Les réponses immunitaires défaillantes à travers de multiples pathologies partagent une tumeur pan-tumeur et circulant cibles lymphocytaires, J Clin Invest, 129 (6): 2463-2479, 2019. doi: 10.1172 / JCI125301
- Mohammed Ali Z, Tokar T , Batruch I, Reid S, Tavares-Brum A, Yip P, Cardinal H, Hébert MJ, Li Y, Kim SJ, Jurisica I , John R, Konvalinka A. Urine Angiotensin II Signature Proteins as Markers of Fibrose in Kidney Transplant Recipients, Transplantation, 103 (6): e146-e158, 2019. doi: 10.1097 / TP.0000000000002676.
- Kaufmann KB, Garcia-Prat L, Liu Q, Ng SWK, Takayanagi SI, Mitchell A, Wienholds E, van Galen P, Cumbaa CA, Tsay MJ, Pastrello C , Wagenblast E, Krivdova G, Minden MD, Lechman ER, Zandi S , Jurisica I , Wang JCY, Xie SZ, Dick JE. Un écran de la souche révèle C3orf54 / INKA1 comme promoteur de la latence des cellules souches de la leucémie humaine, Blood, 133 (20): 2198-2211, 2019. doi: 10.1182 / blood-2018-10-881441
- Mandilaras, V, Garg, S, Cabanero, M, Tan, Q, Pastrello, C , Burnier, J, Karakasis, K, Wang, L, Dhani, NC, Butler, MO, Bedard, PL, Siu, LL, Clarke, B, Shaw, PA, Stockley, T, Jurisica, I , Oza, AM. Mutations TP53 dans le cancer de l'ovaire séreux de haut grade et impact sur les résultats cliniques: une comparaison du séquençage de nouvelle génération et des analyses bioinformatiques. Int J Gyn Cancer , 18 janvier. Pii: ijgc-2018-000087. doi: 10.1136 / ijgc-2018-000087.
- del Toro N, Duesbury M, Koch M, Perfetto L, Shrivastava A, Ochoa D, Wagih O, Piñero J, Kotlyar M, Pastrello C , Beltrao P, Furlong LI, Jurisica I , Hermjakob H, Orchard S, Porras P. Capture impact de la variation sur les interactions moléculaires dans l'ensemble de données sur les mutations du consortium IMEx. Nat Commun, 10 (1): 10, 2019.
- Li L, Guturi KKN, Gautreau B, Patel PS, Saad A, Morii M, Mateo F, Palomero L, Barbour H, Gomez A, Ng D, Kotlyar M, Pastrello C , Jackson HW, Khokha R, Jurisica I , Affar EB , Raught B, Sanchez O, Alaoui-Jamali M, Pujana MA, Hakem A, Hakem R., Ubiquitin ligase RNF8 supprime la signalisation Notch pour réguler le développement mammaire et la tumorigenèse, J Clin Invest , 128 (10): 4525-4542, 2018. doi: 10.1172 / JCI120401
- Kotlyar, M., Pastrello, C., Malik, Z., Jurisica, I. , Mise à jour de l'IID 2018: interactions physiques-protéines physiques spécifiques au contexte chez l'homme, les organismes modèles et les espèces domestiquées. Recherche sur les acides nucléiques, 47 (D1): D581-D589, 2019.
- Endisha, H., Rockel, J., Jurisica, I., Kapoor, M., Le paysage complexe des microARN dans le cartilage articulaire: biologie, pathologie et cibles thérapeutiques, JCI Insight . 3 (17): e121630, 2018.
- Singh, M., Venugopal, C., Tokar, T. , McFarlane, N., Subapanditha, MK, Qazi, M., Bakhshinyan, D., Vora, P., Murty, N., Jurisica, I. , Singh , SK, Ciblage thérapeutique du stade pré-métastatique dans les métastases cérébrales humaines, Cancer Res , 2018. ePub 2018/07/11. DOI: 10.1158 / 0008-5472.CAN-18-1022.
- Wen, B., Tokar, T. , Taibi, A., Chen, J., Jurisica, I. , Comelli, EM Citrobacter rodentium modifie le miRNome colique de la souris, Genes and Immunity , 2018. Sous presse. ePub 2018/05/08. Doi: 10.1038 / s41435-018-0026-z
- Jean-Quartier C, Jeanquartier F, Jurisica I, Holzinger A, In silico cancer research towards 3R. BMC Cancer, 18(1):408, 2018
- Sivade Dumousseau M, Alonso-López D, Ammari M, Bradley G, Campbell NH, Ceol A, Cesareni G, Combe C, De Las Rivas J, Del-Toro N, Heimbach J, Hermjakob H, Jurisica I , Koch M, Licata L, Lovering RC, Lynn DJ, Meldal BHM, Micklem G, Panni S, Porras P, Ricard-Blum S, Roechert B, Salwinski L, Shrivastava A, Sullivan J, Thierry-Mieg N, Yehudi Y, Van Roey K, Orchard S. Nouveaux cas d'utilisation englobants - niveau 3.0 du format HUPO-PSI pour les interactions moléculaires. BMC Bioinformatics, 19 (1): 134, 2018.
- Minatel BC, Martinez VD, Ng KW, Sage AP, Tokar T , Marshall EA, Anderson C, Enfield KSS, Stewart GL, Reis PP, Jurisica I , Lam WL., Découverte à grande échelle de microARN auparavant non détectés spécifiques au foie humain. Hum Genomics, 12 (1): 16, 2018.
- Tokar T , Pastrello C , Ramnarine VR, Zhu CQ, Craddock KJ, Pikor L, Vucic EA, Vary S, Shepherd FA, Tsao MS, Lam WL , Jurisica I Différents microARN exprimés dans l'adénocarcinome pulmonaire inversent les effets des aberrations du nombre de copies des gènes pronostiques . Oncotarget . 9 (10): 9137-9155, 2018.
- Paulitti A, Corallo D, Andreuzzi E, Bizzotto D, Marastoni S, Pellicani R, Tarticchio G, Pastrello C , Jurisica I , Ligresti G, Bucciotti F, Doliana R, Colladel R, Braghetta P, Di Silvestre A, Bressan G, Colombatti A, Bonaldo P, Mongiat M. L'ablation de la protéine matricellulaire EMILIN2 provoque une vascularisation défectueuse due à une production altérée d'IL-8 dépendante de l'EGFR affectant la croissance tumorale, Oncogene, 37 (25): 3399-3414, 2018.
- Tokar, T. , Pastrello, C., Rossos, A., Abovsky, M., Hauschild, AC, Tsay, M., Lu, R., Jurisica, I. mirDIP 4.1 - Base de données intégrative des prévisions de cibles de microARN humain, Nucl Acids Res, D1 (46): D360-D370, 2018.
- Pastrello C, Kotlyar M , Jurisica I. , Utilisation éclairée des données d'interaction protéine-protéine: un focus sur la base de données des interactions intégrées (IID). Méthodes Mol Biol. , 2074: 125-134, 2020. doi: 10.1007 / 978-1-4939-9873-9_10.
- Hauschild, AC , Pastrello, C, Kotlyar, M et Jurisica, I. Données d'interaction protéine-protéine, leur qualité et les principales bases de données publiques. Ed. N. Przulj. Analyzing Network Data in Biology and Medicine, An Interdisciplinary Textbook for Biological, Medical and Computational Scientists, Cambridge University Press, Cambridge, Royaume-Uni, pp.151-192, 2019. ISBN 978-1-108-43223- 8. DOI: 10.1017 / 978110837770
- Wong, S. , Pastrello, C., Kotlyar, M., Faloutsos, C., Jurisica, je . SDREGION: Repérage rapide des communautés changeantes dans les réseaux biologiques. Actes de la conférence internationale ACM SIGKDD sur la découverte des connaissances et l'exploration de données , pp. 867-875, 2018.
- Kotlyar, M., Pastrello, C., Rossos, A., Jurisica, I. Bases de données d'interaction protéine-protéine. Dans: Ranganathan, S., Nakai, K., Schönbach C. et Gribskov, M. (éd.), Encyclopedia of Bioinformatics and Computational Biology , vol. 1 , p. 988–996. Oxford: Elsevier, 2018.
- Rahmati, S., Pastrello, C., Rossos, A., Jurisica, je . Two Decades of Biological Pathway Databases: Results and Challenges, dans: Ranganathan, S., Nakai, K., Schönbach C. et Gribskov, M. (éd.), Encyclopedia of Bioinformatics and Computational Biology , vol. 1 , p. 1071–1084. Oxford: Elsevier, 2018.
- Hauschild, AC , Pastrello, C., Rossos, A., Jurisica, je . Visualization of Biomedical Networks, In: Ranganathan, S., Nakai, K., Schönbach C. and Gribskov, M. (eds.), Encyclopedia of Bioinformatics and Computational Biology , vol. 1 , p. 1016 à 1035. Oxford: Elsevier, 2018.
Autres actualités
Dans d'autres nouvelles, nous avons pu obtenir plusieurs subventions pour permettre le financement du projet, notamment:
- De nouvelles méthodes pour la biologie computationnelle intégrative du Conseil des sciences naturelles et du génie du Canada,
- Cartographie interactome des protéines liées à la maladie à l'aide de la ligature des protéines à médiation par les entéines (SIMPL) de Génome Canada,
- La plateforme de biologie de la signalisation de nouvelle génération des fonds de recherche de l'Ontario
- Instituts de santé du Canada en collaboration avec des agences de financement européennes
Surtout, nous avons également eu la chance d'accueillir deux supporters de World Community Grid et MCM dans notre institut [3]. Dylan Bucci, un élève du lycée Sisler et professeur de réseau et de cybersécurité Robert Esposito a visité notre établissement de recherche pour nous rencontrer, ainsi que nos collaborateurs, les scientifiques qui utilisent les résultats de l'analyse. Il était intéressant pour nous d'en savoir plus sur leur motivation et pour eux de découvrir des liens de recherche directs et indirects avec MCM.
Merci pour toute la puissance de calcul apportée qui rend cette recherche possible.
L'équipe MCM
[1] Rahmati, S., Abovsky, M., Pastrello, C., Jurisica, I. pathDIP: une ressource annotée pour les associations connues et prédites de gènes-voies humaines et l'analyse d'enrichissement de voies. Nucl Acids Res 45 (D1) : D419-D426, 2017.
[2] Rahmati, S., Abovsky, M., Pastrello, C., Kotlyar, M., Lu, R., Cumbaa, CA, Rahman, P., Chandran, V. et Jurisica, I. pathDIP 4: An annotations de voies étendues et ressource d'analyse d'enrichissement pour l'homme, les organismes modèles et les espèces domestiquées, Nucl Acids Res , sous presse. 2019. https://doi.org/10.1093/nar/gkz989[3] Interview de CTV News sur IBM World Community Grid, Mapping Cancer Markers , 3 mai 2019
| By: The Mapping Cancer Markers research team 31 janv. 2020 |
Traduction de la page du site WCG : https://www.worldcommunitygrid.org/about_us/viewNewsArticle.do?articleId=617