 Traduction d'un article de John Bohannon publié sur le site du magazine Wired
Traduction d'un article de John Bohannon publié sur le site du magazine Wired
A peine Zoran Popović avait t-il repéré sa crinière, qu'il reconnu aussitôt David Baker. Aucun doute possible, le visage de Baker est encadré par une large touffe de cheveux bouclés s'organisant en d'imprévisibles spirales qui ne sont pas sans rappeler les protéines complexes qu'il étudie. Ils ne s'étaient jamais rencontrés auparavant, Popović est un expert de la modélisation graphique, un informaticien de l'Université de Washington à Seattle. Baker, quant à lui, est biochimiste dans un laboratoire voisin. David Salesin, un autre informaticien et ami de Baker, a arrangé une rencontre entre les deux scientifiques dans un restaurant à proximité du campus.
Baker a été reconnu comme le meilleur "joueur" du championnat international de chimie des protéines. Lors de cette compétition biennale, les biochimistes reçoivent un sujet de départ, la séquence en acides aminés de plusieurs protéines, ils doivent alors prédire la forme vers laquelle se replient ces protéines. Cette compétition, porte le nom d'expérience communautaire CASP ("Critical Assessment of Techniques for Protein Structure Prediction" : évaluation des techniques de prédiction des structures protéiques).
Baker eu la brillante idée de donner aux utilisateurs la possibilité de diriger l'écran de veille pour tenter des approches spécifiques dont seul l'esprit humain a le secret. Popović répondit négativement de la tête. "Personne ne va faire attention à ça", et il ajouta : "Si tu veux vraiment que les gens s'impliquent, tu dois mettre l'individu au centre".
Salesin pensait la même chose, le repliement des protéines pourrait devenir un jeu vidéo incontournable. C'est la raison pour laquelle il organisa ce déjeuner. Baker, qui avait en tête l'extrême concentration qu'il avait pu déceler dans le regard de son fils de 13 ans jouant aux jeux vidéos, acquiesça également.
Dans le jeu qu'ils imaginent alors, Foldit! (pliez-la!), il n'y a ni orcs, ni quête au marteau anti-gravité. Le jeu se compose d'un entrelacement de spirales multicolores, représentation 3D d'une protéine. Les joueurs utilisent leurs souris pour attraper, plier, tirer et secouer les chaines d'acides aminés dans tous les sens afin de replier la protéine sous sa forme optimale. Les seules règles sont celles de la physique : les charges opposées s'attirent, les liaisons atomiques ont des angles de rotation limités et les sections hydrophiles de la molécule ont tendance à se tourner vers l'extérieur. Plus votre modèle respecte ces règles, et plus vous gagnez des points.
Plus de 100.000 personnes ont téléchargé Foldit depuis l'été dernier, transformant ainsi le jeu en une compétition massivement multijoueur; un origami moléculaire international. Et lorsque ils arrivèrent à des structures presques aussi précises que les structures protéiques du CASP, Baker fît entrer les joueurs dans cette nouvelle compétition.
Quiconque percera les secrets cachés du repliement des protéines ouvrira la voie vers la conception de nouveaux antibiotiques, de nouveaux traitements contre le cancer et même de nouveaux types de biocarburants. Plutôt que de fonder tous ses espoirs sur les ordinateurs pour accélérer sa recherche, Baker tire partie des neurones et de l'irrémédiable envie de jouer de l'Homme. Et si son armée de joueurs acharnés permettait de révéler quelques savants ? Et bien, tant mieux ! répond Baker : "Nous sommes à la recherche de prodiges".
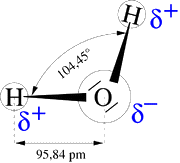
L'hydrogène, la molécule la plus commune dans l'univers, n'est constituée que de deux atomes, un proton et un électron.
Les protéines, les structures chimiques qui sont à la base de tout, des muscles en passant par le lait maternel, se placent à l'exact opposé de l'échelle de la complexité. Constituées de longs méandres de chaînes moléculaires appelées acides aminés, une protéine peut être composée de plus de 10.000 atomes.
La fonction d'une protéine est déterminée par sa structure tridimensionnelle, c'est à dire par la façon dont ses atomes interagissent entre eux. Tenter de rapprocher deux atomes qui se repoussent, c'est comme vouloir réunir les mêmes pôles de deux aimants. Vous pouvez les forcer à se rapprocher, mais la nature préfère suivre la voie qui engendrera le moins de résistance.
 |
Mais plus la molécule est grande, et plus ces combinaisons deviennent complexes. Et les protéines sont très complexes. Bien sûr il existe une autre méthode : envoyer des rayons X sur le cristal d'une protéine peut aider à révéler la position exacte des atomes repliés. Mais cela prend du temps, seule 50.000 structures ont été décryptées depuis la fin des années 1950, alors qu'au cours de ces 10 dernières années, des millions de séquences codant les gènes des protéines ont été découvertes. Pour progresser dans la compréhension de ces protéines, les scientifiques ont besoin d'une approche plus rapide.
Alors pourquoi ne pas utiliser les mathématiques ? Il suffirait de calculer l'énergie de tous les repliements possibles d'une protéine pour mettre en évidence le repliement le plus efficient. Et paf, c'est dans la poche.
 |
|
Le Défi
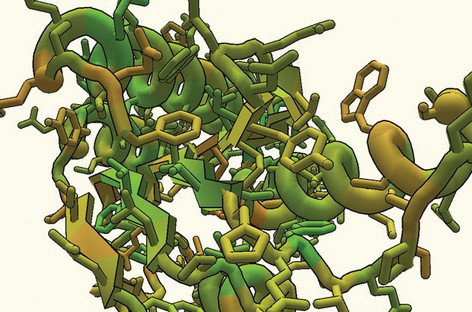
Le jeu en ligne Foldit est conçu pour révêler les mécanismes naturels qui font que des enchevêtrements d'acides aminés (tels que présentés sur cette image) se transforment en protéine. Les joueurs jouent avec leurs souris pour déplacer les pièces comme dans un puzzle. |
Popović a conçu l'interface de Foldit, le jeu permet de modéliser n'importe quelle protéine par une sorte de dessin animé combinant spirales, zigzags, gribouillis et boucles géométriques. Chaque portion de la protéine est mobile, rapprochez deux brins et placez un connecteur scintillant (représentant une liaison hydrogène) pour les plaquer l'un à coté de l'autre. Essayez de placer une boucle dans un espace trop petit pour l'accueillir et des étoiles rouges vous indiqueront les points de collision. Le bouton "wiggle" secoue toute une partie de la structure pour essayer d'améliorer la disposition des "pièces du jeu". C'est le même mécanisme que l'on peut observer lorsque l'on secoue un récipient contenant des pâtes sèches qui auront alors tendance à se concentrer. Ajoutez une fenêtre de Tchat et un indicateur de votre score et vous obtenez un vrai jeu.
Le jour de sa sortie, le 8 mai 2008, un article paru dans la magazine The Economist. L'afflux de joueurs submergea le serveur. Travaillant sur des protéines dont Baker connaissait déjà la structure, les joueurs ("les plieurs") commencèrent rapidement à se faire des amis par l'intermédiaire du canal de discussion intégré au jeu. Ce Tchat leurs permettait d'échanger des idées et des points de sauvegarde; des équipes se sont alors révélées et les efforts collectifs s'avérèrent bien plus efficaces que n'importe quel plieur individuel. Un membre d'une des meilleurs équipes : Jason Kuznicki (pseudonyme : Diderot) lança un wiki que Popović promut en manuel officiel du jeu Foldit. "Nous avons même mis en place un mini Facebook pour eux" précise t-il.
L'atmosphère chaleureuse attire près de 100 nouveaux joueurs par jour, et la compétition acharnée entre les équipes ("Plieurs libres", "Fondation Richard Dawkins", "Plieurs pour Obama",...) encourage tous les joueurs à s'améliorer. Début juin, l'équipe de Baker distribua 5 protéines du CASP à la communauté, et ils croisèrent les doigts.
Une partie mémorable
Le 28 Juillet 2008, aux alentours de minuit, Laurent de Jerphanion (pseudo : Dejerpha) regardait avec incrédulité l'enchevêtrement multicolore s'affichant sur son écran d'ordinateur. Ce responsable marketing de 43 ans vivant à Paris travaillait sur le puzzle T0461 depuis plusieurs nuits. Il ne voyait plus aucune possibilité d'améliorer son jeu, il était proche de la victoire.
ll porta son regard sur le tableau des scores et remarqua qu'un américain de 13 ans, Cheese, avait réussit à le dépasser. Le gamin, de son vrai nom Aristides Poehlman, venait d'améliorer son score de 20 points après avoir effectué un seul mouvement à peine une heure avant la fin de la partie. Mais de Jerphanion n'est pas l'un des meilleurs joueurs de Foldit au monde pour rien. "À nous deux maintenant" grommela t-il.
A l'autre bout du monde, aux environ de 19H00 en Virginie, la famille Poehlman entrait en ébullition. Les parents de Cheese étaient en train de plier sur les ordinateurs à l'étage. A peine son fils venait-il de terminer son coup de maître, qu'Athena, sa mère, lança un message dans la fenêtre de discussion intégrée au jeu : "Wow! Way to go!" ("alors là, chapeau!"). D'autres encouragements ne t'attardèrent pas à affluer en provenance du monde entier de la part du reste de son équipe, "Another Hour, Another Point". Mais durant ce moment d'inattention, Jerphanion venait d'améliorer le meilleur score d'un petit point. C'était un match dantesque.
Poehlman trouva que la forme de sa protéine était parfaite. Trop parfaite, pensa t-il. Pas moyen de faire un nouveau saut de 20 points. Il eu l'idée de reconstruire une boucle d'acides aminés profondément enterrée dans la structure de la protéine, un mouvement risqué. Il se concentrera sur de petits ajustements. Il plaça deux points d'appui sur la chaine principale et il cliqua sur un bouton du jeu pour exécuter un wiggle (secouer) suivi d'un "sidechain shake" (agiter les chaînes latérales de la protéine). Les acides aminés tremblèrent comme un chien mouillé, mais son score ne bougea pas d'un poil.
Pendant ce temps, à Paris, de Jerphanion effectua une rotation de sa protéine pour observer ses entrailles. Une solide amélioration le rendrait imbattable. Il se saisit d'une boucle et donna une petite pichenette pour l'envoyer vers un espace vide mais son petit coup en finesse se transforma en geste de bûcheron. La protéine se transforma en un sapin de noël aggrémenté de guirlandes et de boules de couleur rouge vif, une collision d'acides aminés ! Il annula le mouvement.
Poehlman, lui aussi, tentait d'emprunter un autre chemin. Il tourna la protéine et scruta une boucle brimbalant à l'extrémité de la plus grande hélice. En utilisant la fonction Pull (tirer), il l'accompagna le long du flanc de la protéine et exécuta un nouveau wiggle. Le programme enregistra son score. Poehlman s'empressa d'envoyer un message à son équipe : "je viens de tirer le score d'un point".
Il ne restait qu'une minute de jeu, les parents de Poehlman descendirent pour voir leur fils faire les cents pas devant son ordinateur, rongeant ses ongles jusqu'à l'os. Il avait conscience que Jerphanion pouvait le clouer sur place à tout moment.
Puis la fin de la partie fût sifflée. Poehlman pouvait alors se mettre à guincher et à siffler alors que la structure tridimentionnelle de sa protéine était automatiquement envoyée vers le serveur de Baker.
L'annonce des résultats du CASP
A 9h40 du matin, le microphone crache une annonce : "Y a t-il quelqu'un pour représenter l'équipe de Baker ?". À l'intérieure de la salle de conférence, trois cent scientifiques observent avec attention le moindre mouvement. A l'extérieur, le vent froid de décembre brasse l'air d'un ciel couleur gris ardoise, mais au cœur de l'hôtel Setar Palace près de Cagliari en Sardaigne, l'atmosphère est lourde. Tous les deux ans, la communauté du CASP se rassemble pour assister à l'annonce des résultats.
Les scores globaux viennent juste d'être publiés sur le site internet du CASP, et le Wi-Fi de l'hôtel est mis à rude épreuve par l'auditoire qui essaye d'arracher quelques bribes d'information. Pour donner une note sur chaque casse tête, les juges du CASP utilisent une formule qui compare les estimations par rapport aux données mesurées expérimentalement. Dans la compétition intitulée "tous les coups sont permis", les participants pouvaient utiliser tous les moyens possibles : intelligence, intuition humaine et ordinateurs pour prédire la structure finale des protéines. Le meilleur score est obtenu par l'équipe de Baker dans la catégorie réputée la plus difficile, c'est à dire lorsque les données de départ ressemblent à aucune autre protéine connue. Tout l'auditoir s'attendait déjà à ce résultat. Mais la question qui est sur toutes les lèvres consiste à savoir si les joueurs de Foldit ont contribué aux résultats gagnants.
Deux étudiants en thèse travaillant sous la direction de Baker, James Thompson et Robert Vernon, encore groggys par une nuit d'insomnie et des mois de travail sans relâche sur les protéines du CASP, répondent finalement à la question que tout le monde se pose. Après un bref affrontement avec leur portable qui refuse de fonctionner correctement, ils réussissent finalement à charger leurs résultats. Sur les 15 solutions obtenues par Foldit et soumises par Baker à l'évaluation du CASP, 7 ont terminé dans le haut du classement. L'une des solutions obtenues par Poehlman et ses acolytes a même pris la première place. Une bande de joueurs a réussi à détroner les meilleurs biochimistes.
Cependant, du point de vue de certains, la véritable consécration de Foldit a eu lieu quelques mois auparavant. Les créateurs du jeu ont invité les meilleurs joueurs à Seattle, afin de leurs demander leurs avis pour améliorer l'application. Popović contacta les parents Poehlman. Le chérubin fût complétement abasourdi : "Aristides ne nous croyait pas, il a fallu qu'on lui montre le courriel" raconte sa mère."Le moment de silence dont nous avons eu droit fût désopilant". Poehlman et son père, Louis, ont alors pris l'avion à destination de Seattle. Comme à leur habitude lorsqu'ils sont chez eux, ils ont joué à Foldit pendant des heures dans leur chambre d'hôtel avant de se coucher.
Dans le laboratoire d'informatique de l'Université de Washington, Popović et ses étudiants ont filmé les Poehlmans en train de jouer à Foldit, puis les ont interrogé sur leurs techniques de jeu. Louis était intraitable dans ses analyses et son approche pour chaque puzzle, offrant des justifications sophistiquées à chacun de ses mouvements. Mais lorsque les scientifiques se sont tournés vers Cheese pour lui demander comment il savait qu'il fallait modifier la protéine de telle façon, par exemple orienter les chaines latérales hydrophobes vers le coeur de la protéine, il haussa les épaules et répondit "ça semble juste correct".
Et c'est exactement ce que Baker recherchait : "C'est l'idée que j'avais avancé depuis le départ, j'esperai que Foldit nous aiderait à dénicher des prodigues du repliement des protéines, c'était une bonne prévision. C'est fantastique de voir que le rêve est devenu réalité" claironne Baker.
Le prochain CASP est prévu pour dans deux ans, et Baker ne veut pas perdre l'élan impulsé par Foldit. avec Popović, il a lancé un défi aux joueurs : concevoir une nouvelle protéine. Le laboratoire de Baker développe de nouvelles cibles pour les médicaments contre le cancer, le sida et la maladie d'Alzheimer. Le travail "des plieurs" est de concevoir une petite protéine médicamenteuse avec une structure adéquate et les bonnes propriétés ou caractéristiques de liaison. Ce n'est pas qu'un simple exercice intellectuel. Baker précise qu'il synthésiera les structures les plus promettrices et leur fera subir une batterie de tests en laboratoire. Ces protéines pourraient avoir une valeur thérapeutique dans le monde réel et non plus uniquement virtuellement. Si c'est le cas, les joueurs de Foldit partageront la gloire de la découverte et ce sera la première fois que le meilleur scoreur d'un jeu vidéo deviendra prix Nobel.
John Bohannon
(gonzo at aaas.org) est un correspondant du magazine Science
basé à Vienne en Autriche.


