 Traduction du message de David Baker sur le forum Rosetta.
Traduction du message de David Baker sur le forum Rosetta.
Bienvenue à tous nos nouveaux participants ! La récente augmentation de la puissance de calcul est extrêmement stimulante et nous en faisons bonne utilisation !
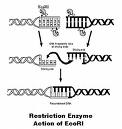
Aujourd'hui (mardi 8 mai), nous avons eu une longue réunion à Seattle avec les trois autres groupes de recherche avec lesquels nous collaborons pour créer des enzymes de restriction de l'ADN pour corriger les mutations responsables de maladies comme je l'ai décrit il y a une ou deux semaines. Nos associés ont examiné une enzyme de restriction de l'ADN que nous avons conçue pour cibler les mutations responsables de l'immunodéficience sévère combinée (SCID) dans les cellules humaines. Les résultats préliminaires indiquent qu'elle vise l'emplacement du SCID juste comme nous l'avions conçu pour ! Les prochaines étapes que nos collaborateurs suivront pour cette maladie ainsi que pour des enzymes de ciblages d'autres maladie sera d'injecter à l'intérieur de l'organisme de souris atteintes de la maladie la version normale du gène (non vecteur de la maladie) et de voir si les mutations sont corrigées et la maladie traitée. Mes étudiants et moi sommes impatients de savoir si nous pouvons traiter les maladies humaines avec des modélisations d'enzymes de restriction de l'ADN mais nos collaborateurs soulignent qu'il devra y avoir beaucoup d'essais.
J'ai promis une explication sur les unités de travail "Search_Pairings". Comme vous le savez peut-être, beaucoup de protéines contiennent des brins bêta qui se regroupent par deux pour former un feuillet bêta. Pour de nombreuses protéines, la clef pour prévoir correctement une structure passe par un regroupement correct des paires de brins de bases. Le problème est que dans certains cas la formation des paires de bases est extrêmement rare. Pour les unités de travail Search_Pairings je résous ce problème par la mise au point d'une liste de toutes les paires possibles, et au début de chacun de vos calculs une de ces paires est choisie aléatoirement et forcée à être présente dans tout le calcul de cette structure. Ce procédé s'assure que toutes ces paires sont prélevées à une fréquence raisonnable pour qu'aucune paires importantes ne soient omises. Si vous obtenez une de ces unités de travail, regardez si vous pouvez sélectionner les paires qui sont maintenues fixe !
J'examine cette approche sur des protéines de structure connue. En parallèle, Rob Vernon, l'un de mes étudiants, utilise la même approche pour établir des modèles pour les chaines formées de protéine alpha-Synucléine qui est impliquée dans la maladie de Parkinson.


