 Traduction du message posté sur le forum du World Community Grid
Traduction du message posté sur le forum du World Community Grid
Bonjour à tous,
Voici un court résumé de la situation que j'ai écrit pour la mise à jour du statut du projet. Il indique brièvement ce qui a été fait jusqu'ici, pourquoi celà a été fait (non, ce n'est pas simplement pour l'amusement de faire d'énormes calculs !) , et vers quel objectif nous nous dirigeons (si tout va bien !).
Je voudrais de nouveau remercier tout ceux d'entre vous qui participent au projet.
Cordialement,
Sophie
Pendant la première phase du projet, des calculs d'arrimage (liaison) moléculaire croisé ayant pour base une banque de données de plus de 150 protéines ont été effectués à l'aide du WCG. C'est-à-dire, que nous avons modelisé chaque combinaison de protéines afin de voir comment elles pouvaient interagir entre elles.
Les données résultant de ces calculs vont maintenant être analysées en fonction de l'énergie de liaison, des zones de fixation (liaison) protéine-protéine et des résidus issus de cette fixation. Ceci en vue de savoir si nous pouvons déterminer les paramètres qui peuvent aider à détecter à l'intérieur de la base de données les associations expérimentales (c'est à dire les protéines qui appartiennent in vivo au même complexe protéine-protéine). Les interfaces de complexes protéine-protéine qui ont été produites pendant la première phase du projet seront analysées pour voir si nos calculs peuvent être utiles pour la prévision des zones d'interaction entre protéines (comme celà a été confirmé par les résultats préliminaires qui ont été obtenus à l'aide d'un ensemble réduit).
Nous utiliserons également les résultats des calculs de liaison moléculaire pour examiner le programme (appelé JET) développé par le groupe d'Alessandra Carbone pour la prévision du site de fixation des protéines à l'aide d'informations évolutionnistes.
Si les résultats sont satisfants (en termes de partenaires et de sites de fixation) nous nous tournerons vers la deuxième phase du projet. Dans cette deuxième phase, nous incorporons JET dans le programme de liaison moléculaire croisée. L'information apportée par JET au sujet du site de fixation de la protéine permettra des calculs d'arrimage moléculaire beaucoup plus efficaces (et donc beaucoup plus rapides) de sorte que nous puissions travailler sur un plus grand ensemble de données (jusqu'à 4000 protéines).
Sur le plus long terme, le but de ce projet est de développer un outil performant qui permettrait de détecter dans une banque de données de protéines (telle que la banque de données des protéines (Protein Data Bank), qui comprends plus de 40 000 protéines) les interactions potentielles associées pour des protéines spécifiques qui sont connues comme étant impliquées dans les maladies neuromusculaires.
Progression du projet
Actuellement, le projet utilise 47% de la puissance de calcul de la plateforme World Community Grid.
A ce rythme, la fin de la première phase du projet est prévue pour dans 32 jours :
Date de fin du projet estimée pour le 26 juin 2007.
Progression du projet sur les 3 dernière semaines
|
|
% des calculs
|
% des protéines
|
|
2 Mai 2007
|
47,51 |
85,1
|
|
9 Mai 2007
|
53,75
|
89,88
|
|
16 Mai 2007
|
61,41
|
94,64
|
|
23 Mai 2007
|
69,36
|
97,02
|
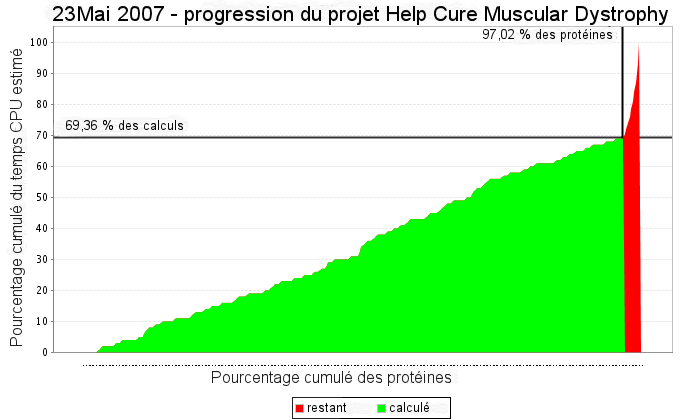
Avancement du projet au 23 Mai 2007
|
Calculées
|
Total
|
|
|
Nombre de protéines
|
163 (97,024%)
|
168
|
|
Temps CPU estimé
|
10 s 32 a 231 j 10 h 34 min 30 s
( 69.36 %) |
14 c 88 a 237 j 19 h 45 min 54 s
|
|
Quantité de données
|
114 Go (90.14 %)
|
126 Go
|


