Traduction du message du Dr. Bonneau sur le forum du World Community Grid
Traduction du message du Dr. Bonneau sur le forum du World Community Grid
Le projet HPF1 (Protéome Humain - phase 1) est terminé depuis plusieurs mois et nous pouvons maintenant annoncer que le premier article relatif aux calculs menés sur la grille vient d'être publié (c'est la thèse de Lars avec une partie que j'ai écrite l'année dernière, mais c'est le premier papier qui sera lu ;-)). Lars, David et moi avons écrit ce papier il y a déjà plusieurs mois mais les nombreuses étapes nécessaires à la publication prennent du temps. Nous pensons que c'est le premier papier issu des calculs sur la grille World Community Grid, et le premier d'une longue série.
 Tout d'abord, merci à tous d'avoir rendu ceci possible
Tout d'abord, merci à tous d'avoir rendu ceci possible 

Voici les points principaux de l'article :
-
Cet article aborde les méthodes qui permettent de combiner les anciennes informations fonctionnelles (qui étaient codées dans une base de données appelée la base de données Gene Ontology- Ontologie de gène) avec nos méthodes de prévision de structure.
-
Afin de déterminer le taux de réussite et développer notre méthode, nous avons tout d'abord choisi d'appliquer notre méthode à la levure, un organisme modèle. Une grande partie des connaissances actuelles en biologie découlent d'études effectuées sur des organismes modèles.
-
Les biologistes à travers le monde ont maintenant accès aux données par l'intermédiaire du yeast resource center (centre de ressources sur la levure). Mike Riffle m'a informé que le site est couramment utilisé (d'importantes statistiques d'utilisation pour ce type de ressources).
Notre expérience avec la levure a été positive, nous devons encore travailler sur quelques détails des algorithmes (dans la façon dont nous intégrons les données de la grille avec d'autres bio-sources, le but étant de maximiser l'utilité apportée aux biologistes) et nous préparons également les résultats relatifs à l'Homme. Le prochain article détaillera les résultats pour 150 protéomes (qui aborderont l'ensemble de l'éventail de la vie). La conclusion de l'article fait référence à ce travail : "L'information contenue dans les structures prévues tirera davantage de puissance avec l'intégration d'autres données telles que des mesures quantitatives de l' ARNm (l'ARN messager), des niveaux d'expression des protéines et des protéines ADN, et les interactions protéine-protéine. De tels ensembles de données sont disponibles pour la levure et plusieurs autres organismes dans le cadre des travaux actuels en génomique fonctionnelle, et l'intégration de ces types de données avec les structures prévues devrait contribuer à l'assignation des fonctions des protéines."
Voici une des meilleures figures de la publication :
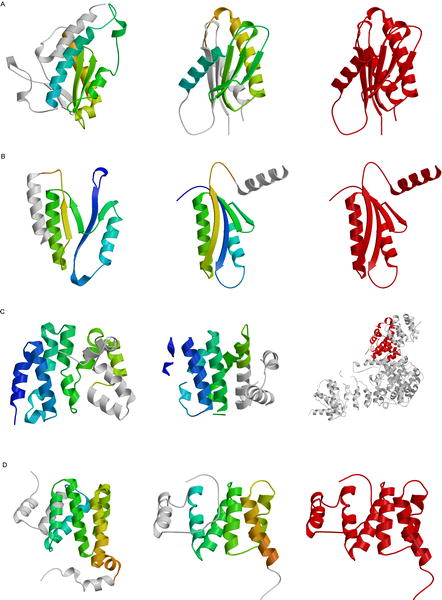
Figure 4 : Prévision des domaines avec le score SCOP d'assignement aux super-familles qui ont été résolus postérieurement.
La structure prévue est illustrée à gauche, et la structure "native" (la forme que la protéine doit avoir pour exercer sa fonction) ou la structure homologue apparait au milieu. La partie qui coordonne le domaine dans la structure résolue est représentée en rouge à droite
Voici le résumé de la publication :
La structure tridimensionnelle d'une protéine peut indiquer beaucoup au sujet des parentés évolutives et des fonctions de cette protéine. Une telle information sur toutes les protéines d'un organisme - le protéome - offrirait une vision plus globale de ces relations, mais résoudre individuellement chaque structure serait une gigantesque tâche. Dans cette étude, nous avons analysé toutes les protéines de saccharomyces cerevisiae pour près de 15 000 domaines disctincts puis nous avons utilisé les méthodes de prévision de structure de novo ainsi que l'informatique répartie pour prévoir les structures pour lesquelles il manquait une similitude d'ordre, par rapport à tous les domaines des protéines dont la structure est connue. Pour surmonter ces incertitudes liées à la prévision de structure de novo, nous avons combiné ces prévisions avec des données (études expérimentales précédentes) relatives à la fonction, la localisation et au processus biologique des protéines pour attribuer les domaines aux familles de protéines. Nos prévisions de domaine du génome et l'assignation de super-familles fournissent la base pour la production d'hypothèses expérimentalement testables au sujet des mécanismes de fonctionnement d'un grand nombre de protéines de la levure.
Appuyer ici pour avoir accès à la totalité de la publication en anglais