Le point sur la recherche par l'équipe du CCN
- Détails
- Écrit par : JeromeC
- Catégorie parente: Actualités
Chers bénévoles, nous avons reçu de l'équipe Smash Childhood Cancer des nouvelles intéressantes sur la recherche que nous aimerions partager avec vous ! Au cours du processus de redémarrage, les chercheurs du SCC ont travaillé d'arrache-pied pour découvrir d'autres moyens de prévenir le cancer chez les enfants, et nous avons hâte de les aider bientôt dans leurs recherches. Voici ce qu'ils nous ont partagé :
« La β-caténine (CTNNB1) est une partie importante de la voie de signalisation Wnt/β-caténine, qui est impliquée dans la régulation du développement embryonnaire et le maintien des organes et des tissus. L'activation de la cascade de signalisation Wnt/β-caténine inhibe la dégradation de la β-caténine, qui à son tour stimule la transcription des gènes cibles en aval.
Une activation anormale de la signalisation β-caténine est observée dans > 70 % des cancers colorectaux et plusieurs tumeurs pédiatriques telles que l'hépatoblastome et le craniopharyngiome. La β-caténine activée favorise la prolifération, la survie et la migration des cellules cancéreuses.
Par conséquent, des stratégies de ciblage de la β-caténine ont été tentées par les chercheurs, mais aucune option efficace n'est encore disponible. L'identification d'inhibiteurs spécifiques contre les domaines de liaison aux protéines β-caténine est l'un de nos projets dans le cadre du programme Smash Childhood Cancers. Tyuji Hoshino a synthétisé plus de 150 composés chimiques ciblant les sites de liaison de la β-caténine sur la base des informations générées par le vaste réseau informatique donné par les volontaires. Ching Lau et Godfrey Chan ont vérifié les propriétés in vitro et in vivoactivités de certains des nouveaux composés générés, avec l'intention d'une éventuelle traduction clinique à l'avenir.
Merci de nous lire !
L'équipe de World Community Grid
Source : https://www.worldcommunitygrid.org/about_us/article.s?articleId=774
- Affichages : 1137
Le vaccin COVID-19 avec des nanoparticules IPD obtient l'approbation complète à l'étranger
- Détails
- Écrit par : JeromeC
- Catégorie parente: Actualités
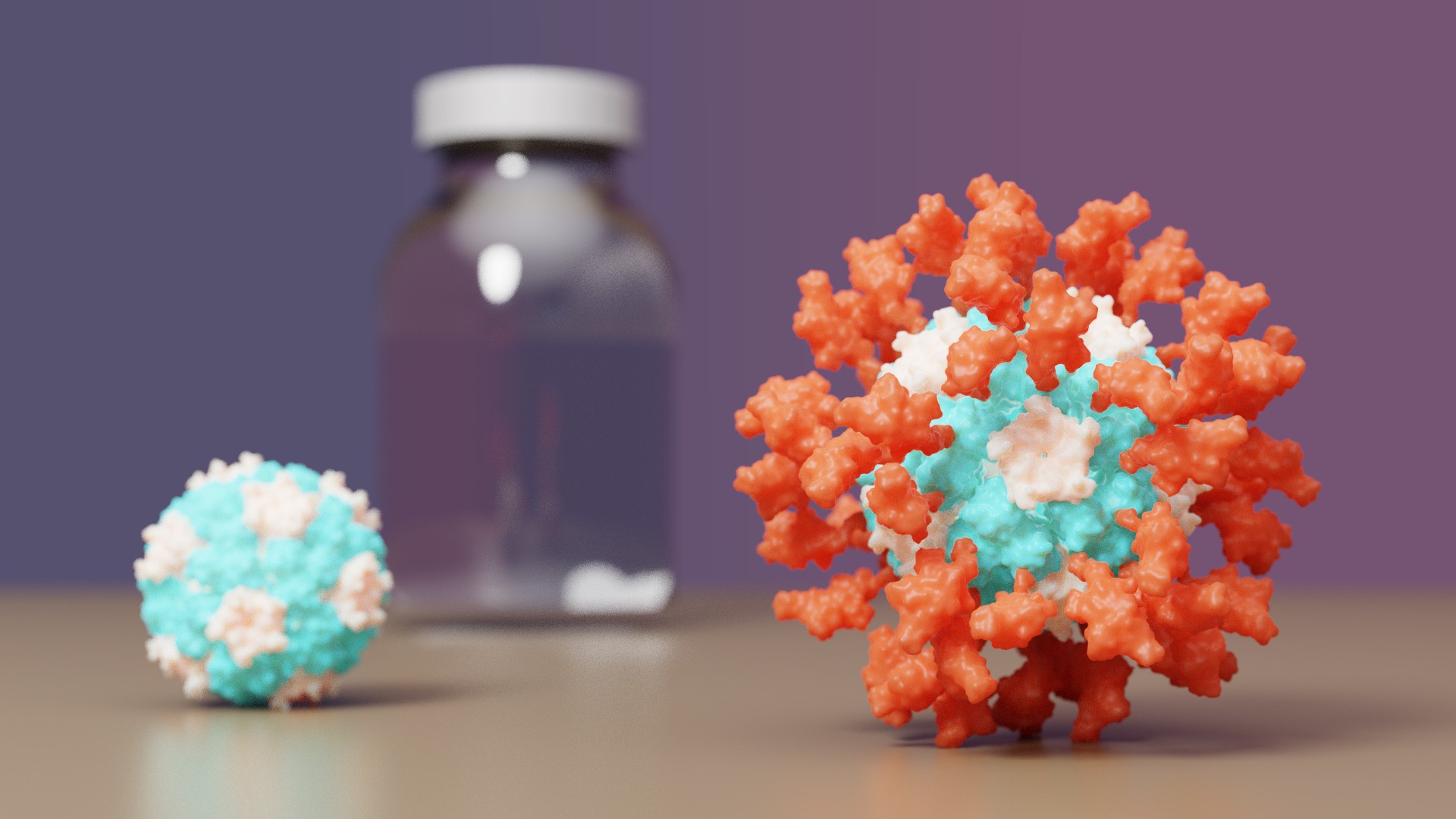
L'IPD est heureux d'annoncer que son premier médicament protéique conçu a été approuvé à l'étranger.
Félicitations et merci à tous les contributeurs de Rosetta@home ! Les calculs que vous avez fournis ont grandement contribué à relever les défis de la conception de protéines de novo, tels que le développement de vaccins, ce qui a permis de réaliser des percées comme celle-ci.
Pour plus d'informations, vous pouvez consulter l'article sur les vaccins de l'IPD.
Une vidéo est également disponible ici.
Extrait du site d'information de l'IPD :
- Les tests cliniques ont montré que le vaccin surpasse celui d'Oxford/AstraZeneca.
- Le vaccin à base de protéines, désormais appelé SKYCovione, ne nécessite pas de congélation.
- L'Université de Washington renonce aux redevances pour la durée de la pandémie.
- La Corée du Sud va acheter 10 millions de doses pour un usage domestique
- Affichages : 1465
WCG : Mise à jour de l'équipe technique de juillet 2022
- Détails
- Écrit par : JeromeC
- Catégorie parente: Actualités

Discussion sur les erreurs actuelles de notre plateforme BOINC et de ce qui est en train d'être corrigé.
Publié le 13/07/2022
Chers volontaires,
Au cours de la semaine dernière, nous sommes entrés dans une nouvelle étape de notre phase de test. Nous avons donné quelques unités de travail à nos volontaires pour qu'ils les crunchent. L'objectif était de voir si des erreurs imprévues se produisaient, ce que nous avons pu trouver grâce aux commentaires de nos volontaires. Si vous avez des questions spécifiques sur les unités de travail limitées, consultez ce fil de discussion sur nos forums.
Voici une mise à jour rapide de notre équipe technique :
Nous avons travaillé pour évaluer la façon dont le système répond à la charge et si la configuration est correcte. Comme beaucoup l'ont remarqué sur les forums, il y a quelques problèmes avec la configuration du backend BOINC. Les utilisateurs auront remarqué des notifications anormales dans le gestionnaire BOINC à propos d'URLs de projet incorrectes et de double rattachement au même projet. Nous avons résolu le problème sous-jacent et nous nous excusons pour la gêne occasionnée.
En outre, nous travaillons à résoudre le manque de synchronisation entre la page de profil du site Web et le gestionnaire BOINC. Nous travaillons également à la résolution d'un problème de blocage avec les unités de travail GPU pour Open Pandemics. Une fois ce problème résolu, nous devrions être en mesure d'augmenter la quantité d'unités de travail que nous pouvons envoyer aux volontaires.
Notre arriéré de problèmes non bloquants a continué de croître au fur et à mesure que nous observons la charge du système, mais dans l'ensemble, nous nous approchons d'un état stable et nous sommes impatients de mettre à jour les utilisateurs avec des statistiques et plus de détails au fur et à mesure que nous continuons le déploiement de notre système de production.
Merci de votre compréhension et de votre soutien continu.
Sincèrement,
L'équipe de World Community Grid
Source : https://www.worldcommunitygrid.org/about_us/article.s?articleId=773
- Affichages : 1534
Lire la suite : WCG : Mise à jour de l'équipe technique de juillet 2022
Les techniques de Help Stop TB (World Community Grid)
- Détails
- Écrit par : JeromeC
- Catégorie parente: Actualités

Les scientifiques de Help Stop TB nous donnent un aperçu des algorithmes utilisés pour le projet, et les raisons de leur choix.
Matériel, système d'exploitation, logiciel, langues utilisées pour #HelpStopTB.
Dans le cadre de notre projet, nous utilisons les serveurs WCG aux côtés de nos machines locales. Ces petites centrales électriques (toutes nommées d'après des icônes australiennes !) fonctionnent sous Linux et ont chacune entre 10 et 20 cœurs et 64 à 128 Go de RAM. La plupart des calculs scientifiques se déroulent désormais sur Linux, et cela est particulièrement important pour nous car nous en avons besoin pour travailler avec GROMACS, le logiciel de dynamique moléculaire que nous utilisons pour simuler les acides mycoliques sur le WCG. Beaucoup de nos scripts sont codés en Perl ou en Python, et nous sommes sur un processus lent de transfert de plus de processus vers Python. Python s'est avéré très utile pour les analyses récentes - en raison de sa nature et de sa popularité open source, il existe de nombreux supports pour développer les outils sur mesure et la formation nécessaires pour les données sur l'acide mycolique.
Certaines des techniques que nous utilisons.
D'après la taille des données et les efforts de l'équipe jusqu'à présent, il est clair que bon nombre des techniques que vous utiliseriez traditionnellement pour étudier la dynamique moléculaire ne sont pas adaptées à un ensemble de données aussi vaste que le nôtre (en particulier avec des molécules aussi souple que nos acides mycoliques !). Pour analyser les données, nous devons utiliser des algorithmes d'apprentissage automatique capables de nous dire pourquoi les acides mycoliques se replient dans certaines formes, mais ce n'est pas aussi simple que de lancer n'importe quelle vieille IA sur le problème. Pour extraire ces informations de pliage cruciales des données, nous avons besoin d'outils d'IA explicables sans les limitations de la boîte noire de nombreuses approches. C'est là qu'intervient notre nouveau membre de l'équipe, Connor : avec une expérience en IA, en calcul scientifique et en développement de méthodes,
Nous avons aussi eu des succès locaux ! Nous avions un peu l'intuition que nous examinions des données non linéaires, et nous avons donc commencé à utiliser une technique d'apprentissage compétitive appelée cartes auto-organisées qui agissent comme une alternative non linéaire aux méthodes linéaires moins efficaces. De plus, nous avons utilisé le travail mathématique de Lyapunov (dont la femme est malheureusement décédée de la tuberculose en 1918) pour commencer à comprendre encore plus en détail les propriétés cachées des acides mycoliques. La prochaine étape avec ces approches consiste à augmenter l'échelle de nos systèmes de test à l'ensemble de données complet. Malheureusement, cela prend du temps à développer et à mettre en œuvre sur quelque chose de cette taille, mais nous continuons à faire des progrès constants.
Source : https://www.worldcommunitygrid.org/news/0419
- Affichages : 1197
Des nouvelles du projet Quchempedia (Quantum Chemistry encycloPed and Intelligence Artificielle)
- Détails
- Écrit par : JeromeC
- Catégorie parente: Actualités

Nouvelles du projet Quchempedia (Quantum Chemistry encycloPed and Intelligence Artificielle.). Ce projet BOINC est porté par l'Université d'Angers.
Dans le cadre des conférences Molecular Modeling and Drug Discovery (M2D2), organisées par le laboratoire Valence Discovery, Thomas Cauchy a fait une présentation de nos travaux. Cette vidéo est destinée aux chercheurs mais Thomas fait, comme toujours, un effort important pour vulgariser le sujet. J'espère que vous apprécierez !
Lien du live vidéo (en anglais) : https://youtu.be/DLaJ529c1ag
Résumé : Au-delà de la recherche active de nouveaux médicaments, les méthodes de génération de novo sont également une formidable opportunité pour la découverte de matériaux moléculaires. Cependant, l'espace chimique de ces matériaux diffère de celui des molécules bioactives. Cette conférence présentera les défis inhérents à ce genre de problèmes. Pour la plupart des matériaux moléculaires, les nouvelles cibles doivent avoir des propriétés électroniques spécifiques. Cela signifie normalement une évaluation très coûteuse par des calculs de mécanique quantique. De plus cette évaluation nécessite une connaissance des positions atomiques en trois dimensions. Toutes ces contraintes spécifiques nous ont amenés à proposer notre propre méthode de génération basée sur EvoMol, un algorithme évolutif efficace. Libre de parcourir tout l'espace chimique, les méthodes qui limitent les solutions à des molécules réalistes seront présentées.
Source : https://quchempedia.univ-angers.fr/athome/forum_thread.php?id=173#1717
- Affichages : 1230
Mise à jour du projet MLC du printemps 2022 : édition complète de DS2 !
- Détails
- Écrit par : JeromeC
- Catégorie parente: Actualités

Cela fait un moment que nous n'avons pas posté de mise à jour, mais cela ne veut pas dire que le projet est resté inactif ! Si vous avez suivi notre serveur Discord, vous savez que nous avons continué à progresser, et grâce à nos bénévoles, aujourd'hui est un jour de fête !
Voici un résumé de l'état actuel du projet :
Résumé
- Le calcul de DS2 est terminé ! Au 1er avril 2022, nous avons enfin franchi le seuil des 10 000 réseaux formés pour ParityModified, achevant ainsi notre calcul pour DS2. Cela a pris beaucoup de temps, et l'ensemble complet de données devrait aider les chercheurs à comprendre comment les réseaux neuronaux encodent les données.
- Tous les tarballs DS1/DS2 peuvent être téléchargés à partir de https://www.mlcathome.org/mlds.html. C'est votre travail, et maintenant il est libre pour vous ou n'importe qui d'autre de l'étudier et de le développer !
- Les archives de DS3 sont toujours en attente. Les calculs pour DS3 ont été achevés l'année dernière, mais nous n'avons pas encore téléchargé les ensembles de données complets sur le site Web. Nous nous sommes concentrés sur l'analyse, et la taille même de l'ensemble de données peut causer des maux de tête, rendant l'empaquetage une tâche fastidieuse. Nous les publierons ici dès qu'ils seront disponibles.
- Les WUs de DS4 sont sortis ! Les WUs DS4 sont sorties pour notre client CPU, et les progrès ont commencé là. DS4 est beaucoup plus compliqué à gérer en arrière-plan parce qu'il a plusieurs ensembles de formation qui ont des exigences différentes, mais nous poussons de nouvelles WUs aussi vite que nous le pouvons.
- Nous mettons en pause les WUs GPU : Cela nous attriste, mais nous n'avons pas réussi à mettre à jour nos clients GPU pour prendre en charge les WU DS4. Et comme nous nous concentrons sur l'analyse des résultats que nous avons, nous avons de moins en moins de temps pour nous concentrer sur le développement de clients autres que le client CPU. Lorsque la file d'attente GPU actuelle sera épuisée, nous n'enverrons plus de travail GPU jusqu'à ce que nous ayons le temps de redonner la priorité au portage d'un client GPU. La maintenance d'un client GPU a pris beaucoup plus de temps et d'efforts que prévu, et à moins que nous puissions obtenir une aide extérieure, cela restera une faible priorité pour le moment. Nous apprécions vraiment nos volontaires GPU, mais pour le moment nous n'avons pas de travail à envoyer, et nous vous encourageons à tourner votre matériel pour soutenir d'autres projets valables qui peuvent supporter votre matériel !
- Nous explorons le portage du client CPU vers Rust. De plus, notre dépendance à PyTorch est plus devenue une entrave à la portabilité qu'un atout. Bien que l'écosystème des réseaux neuronaux dans Rust soit loin d'être aussi robuste, la capacité de Rust à compiler un binaire statique ciblant un grand nombre d'architectures et de systèmes d'exploitation est très attrayante pour la portabilité. En tant que tel, nous cherchons à porter notre client MLC CPU en pur rust, avec une option pour supporter les GPUs à partir de la même base de code dans le futur. Si vous connaissez Rust et êtes intéressé, veuillez contacter les administrateurs de MLC.
Veuillez noter qu'il y a encore des WUs DS2 dans la file d'attente de travail, nous vous demandons de continuer à les cruncher, car il est toujours préférable d'avoir plus d'échantillons en réserve. Cependant, nous ne prévoyons pas de mettre en file d'attente d'autres UWs DS1/2/3, et toutes les nouvelles UWs ajoutées seront DS4 ou plus. Ceci s'applique également à la file d'attente des GPU.
Nous sommes très enthousiastes quant aux WUs DS4 à venir, et cela devrait nous aider à démontrer notre théorie selon laquelle des réseaux similaires se regroupent dans l'espace des paramètres à la fois dans les réseaux feed forward et CNN, ainsi que dans les RNN utilisés dans DS1/2/3. Au-delà de DS4, nous avons quelques idées mais rien de concret pour le moment. Nous vous tiendrons informés au fur et à mesure que nous avancerons.
Merci encore à tous nos bénévoles qui soutiennent le projet et aident la science.
-- Les administrateurs de MLC@Home
Page d'acceuil : https://www.mlcathome.org/
Invitation discord : https://discord.gg/BdE4PGpX2y
Twitter : @MLCHome2
Source : https://www.mlcathome.org/mlcathome/forum_thread.php?id=267&postid=1492#1492
- Affichages : 1419
- SiDock : statut d'avancement du project - mars 2022
- Le transfert des données de WCG est en cours...
- OpenPandemics - COVID-19 : Mise à jour de février
- L'intégration d'OONI Probe par iThena renforce la couverture des mesures de censure dans le monde entier
- WCG - OpenPandemics - COVID-19 : Mise à jour de septembre
- WCG - Help Stop TB : Mise à jour de septembre
Page 4 sur 35



